+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Central part (C10) of bacteriophage SU10 capsid | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Protein of unknown function DUF5309 / Family of unknown function (DUF5309) / Major head protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Escherichia phage vB_EcoP_SU10 (ファージ) Escherichia phage vB_EcoP_SU10 (ファージ) | |||||||||
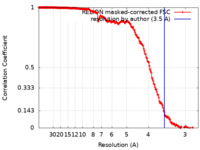
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Siborova M / Fuzik T / Prochazkova M / Novacek J / Plevka P | |||||||||
| 資金援助 |  チェコ, 1件 チェコ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Tail proteins of phage SU10 reorganize into the nozzle for genome delivery. 著者: Marta Šiborová / Tibor Füzik / Michaela Procházková / Jiří Nováček / Martin Benešík / Anders S Nilsson / Pavel Plevka /   要旨: Escherichia coli phage SU10 belongs to the genus Kuravirus from the class Caudoviricetes of phages with short non-contractile tails. In contrast to other short-tailed phages, the tails of Kuraviruses ...Escherichia coli phage SU10 belongs to the genus Kuravirus from the class Caudoviricetes of phages with short non-contractile tails. In contrast to other short-tailed phages, the tails of Kuraviruses elongate upon cell attachment. Here we show that the virion of SU10 has a prolate head, containing genome and ejection proteins, and a tail, which is formed of portal, adaptor, nozzle, and tail needle proteins and decorated with long and short fibers. The binding of the long tail fibers to the receptors in the outer bacterial membrane induces the straightening of nozzle proteins and rotation of short tail fibers. After the re-arrangement, the nozzle proteins and short tail fibers alternate to form a nozzle that extends the tail by 28 nm. Subsequently, the tail needle detaches from the nozzle proteins and five types of ejection proteins are released from the SU10 head. The nozzle with the putative extension formed by the ejection proteins enables the delivery of the SU10 genome into the bacterial cytoplasm. It is likely that this mechanism of genome delivery, involving the formation of the tail nozzle, is employed by all Kuraviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14484.map.gz emd_14484.map.gz | 88.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14484-v30.xml emd-14484-v30.xml emd-14484.xml emd-14484.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14484_fsc.xml emd_14484_fsc.xml | 18 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14484.png emd_14484.png | 268.3 KB | ||
| その他 |  emd_14484_half_map_1.map.gz emd_14484_half_map_1.map.gz emd_14484_half_map_2.map.gz emd_14484_half_map_2.map.gz | 407 MB 407.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14484 http://ftp.pdbj.org/pub/emdb/structures/EMD-14484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14484 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7z45MC  7z44C  7z46C  7z47C  7z48C  7z49C  7z4aC  7z4bC  7z4fC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14484.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14484.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.38 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_14484_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
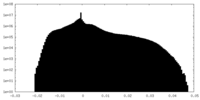
| 投影像・断面図 |
| ||||||||||||
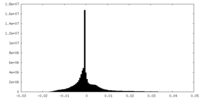
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14484_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
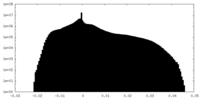
| 投影像・断面図 |
| ||||||||||||
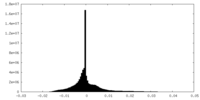
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia phage vB_EcoP_SU10
| 全体 | 名称:  Escherichia phage vB_EcoP_SU10 (ファージ) Escherichia phage vB_EcoP_SU10 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia phage vB_EcoP_SU10
| 超分子 | 名称: Escherichia phage vB_EcoP_SU10 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 1519788 / 生物種: Escherichia phage vB_EcoP_SU10 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 9.257 MDa |
-分子 #1: Major head protein
| 分子 | 名称: Major head protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage vB_EcoP_SU10 (ファージ) Escherichia phage vB_EcoP_SU10 (ファージ) |
| 分子量 | 理論値: 38.616457 KDa |
| 配列 | 文字列: MANPTLFVSY DQNGKKLSFA NWISVLSPQD TPFVSMTGKE SINQTIFSWQ TDALASVDGN NAHVEGSRAE DGEMKPTVIK SNVTQILRK VVRVSDTANT TANYGRGREL MYQLEKKGKE IKRDLEKILL SGQARTDVLA DQYLTNSAAD PAVAGLNDTH A ARKTGAFQ ...文字列: MANPTLFVSY DQNGKKLSFA NWISVLSPQD TPFVSMTGKE SINQTIFSWQ TDALASVDGN NAHVEGSRAE DGEMKPTVIK SNVTQILRK VVRVSDTANT TANYGRGREL MYQLEKKGKE IKRDLEKILL SGQARTDVLA DQYLTNSAAD PAVAGLNDTH A ARKTGAFQ FLCAHGGLAG GVVDKTKNGP ADPDTGAVTV KVAQNASNPT TNIGFDEADI FDMTLQLYTA GSEADIIMIN PA HAKIFAG LQENTQGSRK RIFENTKQFI YEVNSITDPL GQSYKIIVNR WMPTDAVYFF RSADWTQMVL RAPKRTELAK DGS YEKWMI EMEVGLRHRN PYASGVLFTA AGKAAA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | PFU 10^11 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 59000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 49.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7z45: |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X