+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | STRUCTURE OF THE HUMAN INNER KINETOCHORE CCAN COMPLEX AT 10A RESOLUTION | |||||||||
 マップデータ マップデータ | main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | INNER KINETOCHORE / CCAN /  COMPLEX / COMPLEX /  DNA BINDING PROTEIN (DNA結合タンパク質) / DNA BINDING PROTEIN (DNA結合タンパク質) /  CELL CYCLE (細胞周期) CELL CYCLE (細胞周期) | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
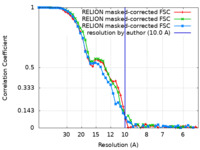
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 10.0 Å クライオ電子顕微鏡法 / 解像度: 10.0 Å | |||||||||
 データ登録者 データ登録者 | Vetter IR / Pesenti M / Raisch T | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: Structure of the human inner kinetochore CCAN complex and its significance for human centromere organization. 著者: Marion E Pesenti / Tobias Raisch / Duccio Conti / Kai Walstein / Ingrid Hoffmann / Dorothee Vogt / Daniel Prumbaum / Ingrid R Vetter / Stefan Raunser / Andrea Musacchio /  要旨: Centromeres are specialized chromosome loci that seed the kinetochore, a large protein complex that effects chromosome segregation. A 16-subunit complex, the constitutive centromere associated ...Centromeres are specialized chromosome loci that seed the kinetochore, a large protein complex that effects chromosome segregation. A 16-subunit complex, the constitutive centromere associated network (CCAN), connects between the specialized centromeric chromatin, marked by the histone H3 variant CENP-A, and the spindle-binding moiety of the kinetochore. Here, we report a cryo-electron microscopy structure of human CCAN. We highlight unique features such as the pseudo GTPase CENP-M and report how a crucial CENP-C motif binds the CENP-LN complex. The CCAN structure has implications for the mechanism of specific recognition of the CENP-A nucleosome. A model consistent with our structure depicts the CENP-C-bound nucleosome as connected to the CCAN through extended, flexible regions of CENP-C. An alternative model identifies both CENP-C and CENP-N as specificity determinants but requires CENP-N to bind CENP-A in a mode distinct from the classical nucleosome octamer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
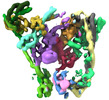
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14099.map.gz emd_14099.map.gz | 7.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14099-v30.xml emd-14099-v30.xml emd-14099.xml emd-14099.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14099_fsc.xml emd_14099_fsc.xml emd_14099_fsc_2.xml emd_14099_fsc_2.xml emd_14099_fsc_3.xml emd_14099_fsc_3.xml | 4.7 KB 4.7 KB 4.7 KB | 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14099.png emd_14099.png | 159.9 KB | ||
| マスクデータ |  emd_14099_msk_1.map emd_14099_msk_1.map emd_14099_msk_2.map emd_14099_msk_2.map emd_14099_msk_3.map emd_14099_msk_3.map | 8 MB 8 MB 8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14099.cif.gz emd-14099.cif.gz | 4.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14099 http://ftp.pdbj.org/pub/emdb/structures/EMD-14099 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14099 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14099 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14099.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14099.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.72 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14099_msk_1.map emd_14099_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_14099_msk_2.map emd_14099_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #3
| ファイル |  emd_14099_msk_3.map emd_14099_msk_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human inner kinetochore CCAN complex
| 全体 | 名称: human inner kinetochore CCAN complex |
|---|---|
| 要素 |
|
-超分子 #1: human inner kinetochore CCAN complex
| 超分子 | 名称: human inner kinetochore CCAN complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#15 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 451 KDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 6.8 / 詳細: 0.0025% Triton |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 286 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 Bright-field microscopy / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / 球面収差補正装置: Krios with CS corrector / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 14 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 55.8 e/Å2 詳細: 2678 images were collected in movie-mode with 60 frames per image on Gatan K3 in superresolution mode with 0.34 A pixel size / native pixel size 0.68A |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X