+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM of Aedes Aegypti Toll5A dimer bound to Spz1C | |||||||||
 マップデータ マップデータ | EM map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Aedes aegypti (ネッタイシマカ) Aedes aegypti (ネッタイシマカ) | |||||||||
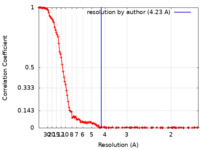
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.23 Å クライオ電子顕微鏡法 / 解像度: 4.23 Å | |||||||||
 データ登録者 データ登録者 | Gangloff M / Hardwick SW / Chirgadze DY | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure and dynamics of Toll immunoreceptor activation in the mosquito Aedes aegypti. 著者: Yoann Saucereau / Thomas H Wilson / Matthew C K Tang / Martin C Moncrieffe / Steven W Hardwick / Dimitri Y Chirgadze / Sandro G Soares / Maria Jose Marcaida / Nicholas J Gay / Monique Gangloff /   要旨: Aedes aegypti has evolved to become an efficient vector for arboviruses but the mechanisms of host-pathogen tolerance are unknown. Immunoreceptor Toll and its ligand Spaetzle have undergone ...Aedes aegypti has evolved to become an efficient vector for arboviruses but the mechanisms of host-pathogen tolerance are unknown. Immunoreceptor Toll and its ligand Spaetzle have undergone duplication which may allow neofunctionalization and adaptation. Here we present cryo-EM structures and biophysical characterisation of low affinity Toll5A complexes that display transient but specific interactions with Spaetzle1C, forming asymmetric complexes, with only one ligand clearly resolved. Loop structures of Spaetzle1C and Toll5A intercalate, temporarily bridging the receptor C-termini to promote signalling. By contrast unbound receptors form head-to-head homodimers that keep the juxtamembrane regions far apart in an inactive conformation. Interestingly the transcriptional signature of Spaetzle1C differs from other Spaetzle cytokines and controls genes involved in innate immunity, metabolism and tissue regeneration. Taken together our results explain how upregulation of Spaetzle1C in the midgut and Toll5A in the salivary gland shape the concomitant immune response. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11982.map.gz emd_11982.map.gz | 103.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11982-v30.xml emd-11982-v30.xml emd-11982.xml emd-11982.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11982_fsc.xml emd_11982_fsc.xml | 17.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11982.png emd_11982.png | 117.3 KB | ||
| その他 |  emd_11982_half_map_1.map.gz emd_11982_half_map_1.map.gz emd_11982_half_map_2.map.gz emd_11982_half_map_2.map.gz | 194.2 MB 194.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11982 http://ftp.pdbj.org/pub/emdb/structures/EMD-11982 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11982 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11982 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11982.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11982.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map
| ファイル | emd_11982_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
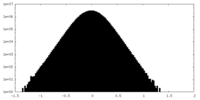
| 投影像・断面図 |
| ||||||||||||
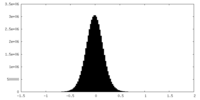
| 密度ヒストグラム |
-ハーフマップ: Half map
| ファイル | emd_11982_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
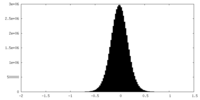
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM of Aedes Aegypti Toll5A dimer bound to Spz1C
| 全体 | 名称: Cryo-EM of Aedes Aegypti Toll5A dimer bound to Spz1C |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM of Aedes Aegypti Toll5A dimer bound to Spz1C
| 超分子 | 名称: Cryo-EM of Aedes Aegypti Toll5A dimer bound to Spz1C タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 分子量 | 理論値: 230 KDa |
-超分子 #2: Toll-like receptor
| 超分子 | 名称: Toll-like receptor / タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Aedes aegypti (ネッタイシマカ) Aedes aegypti (ネッタイシマカ) |
-超分子 #3: AAEL013433-PA
| 超分子 | 名称: AAEL013433-PA / タイプ: complex / ID: 3 / キメラ: Yes / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Aedes aegypti (ネッタイシマカ) Aedes aegypti (ネッタイシマカ) |
-分子 #1: Toll-like receptor
| 分子 | 名称: Toll-like receptor / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Aedes aegypti (ネッタイシマカ) Aedes aegypti (ネッタイシマカ) |
| 分子量 | 理論値: 87.675773 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TSTKRFTCPE ESEASNCSCE EFPSKTHFYC PDFNPTLYVD VEDRMRVDFK CYDEPHDFKS LPNLAIGSVK LLTVVDCVLD DDRPILESF KFLEVADVRS FVYNNHENGI RYNAKYFEGM EQLENLTLAR GVVSIDRDTF SGFLNLKRLT IEHNKLNLQP G TFEALSNL ...文字列: TSTKRFTCPE ESEASNCSCE EFPSKTHFYC PDFNPTLYVD VEDRMRVDFK CYDEPHDFKS LPNLAIGSVK LLTVVDCVLD DDRPILESF KFLEVADVRS FVYNNHENGI RYNAKYFEGM EQLENLTLAR GVVSIDRDTF SGFLNLKRLT IEHNKLNLQP G TFEALSNL TYLGLVYNGL NEIQPGLFDG LESLEALSLS YNDIKSLSAG SFNGLSSLRM LNLRVNKIES FDANTFASLK EL SRLEITL NPFVSLPRGL FSENKKLKTL ILTNNRKLVT LPEELLANLK ELTVVNLSHN GVGNLPESLL SGSSGIIELN LGY NRLNSL PEELLSDQPQ LQVLNLDHNQ LESIPDYFLE RNVELQTLYL SHNRLRSLSE KAFTKLKNLK ELHLENNQLQ TIPQ FLFSG TPKLEEIYMQ NNQLALHANS FINEELSIAD NDNTPFQVLQ KLRILHLRNN SISTIFQDWY INNLEMQSLD LSFNK LPGL SYTQLQFQSN ITLNLSNNEI SQVLLIDDLD LQPYQRINVD LNHNPLNCNC NALKFIQLIQ SKAEHGLQFN VDQLRC SEP PNLLDATMDQ LQTKDLLCDF ESADDCPKDC QCAMRLLDHT VIVNCSGRGL TEFPDLPIPS QLHEDFNALE VHVENNR LT KLPNLTKHNE ITQLYARNNS IQNLLPHNIP SKLRIIDLSQ NLLKMIDDST LAQINRSSHL ETIRLSQNQW LCDCPASS F LIFVQQNSRL ISDMSAIRCH PSGKSLDSIT VNELCFEDYT TENLYFQ |
-分子 #2: AAEL013433-PA
| 分子 | 名称: AAEL013433-PA / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Aedes aegypti (ネッタイシマカ) Aedes aegypti (ネッタイシマカ) |
| 分子量 | 理論値: 12.941044 KDa |
| 組換発現 | 生物種:   Baculovirus expression vector pFastBac1-HM (バキュロウイルス科) Baculovirus expression vector pFastBac1-HM (バキュロウイルス科) |
| 配列 | 文字列: SDTANAPFLC ESEQLLIHPK EELSRNNSMV WIVNTKDYKQ GVRIEKCLKR QLGKPCNFCD ADTECKQLFH YRTLVAVDKV TKKPYKEQV LLPSCCKCAK ILSTGWSHPQ FEK |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK I / 詳細: Blotting force 0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 2.0 sec. / 平均電子線量: 51.1 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X