+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11605 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Respiratory chain supercomplex I1III2IV1 in rat heart mitochondria | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | |||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 29.6 Å クライオ電子顕微鏡法 / 解像度: 29.6 Å | |||||||||
 データ登録者 データ登録者 | Chesnokov YM / Kamyshinsky RA | |||||||||
| 資金援助 |  ロシア, 1件 ロシア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2021 ジャーナル: Int J Mol Sci / 年: 2021タイトル: Ordered Clusters of the Complete Oxidative Phosphorylation System in Cardiac Mitochondria. 著者: Semen Nesterov / Yury Chesnokov / Roman Kamyshinsky / Alisa Panteleeva / Konstantin Lyamzaev / Raif Vasilov / Lev Yaguzhinsky /  要旨: The existence of a complete oxidative phosphorylation system (OXPHOS) supercomplex including both electron transport system and ATP synthases has long been assumed based on functional evidence. ...The existence of a complete oxidative phosphorylation system (OXPHOS) supercomplex including both electron transport system and ATP synthases has long been assumed based on functional evidence. However, no structural confirmation of the docking between ATP synthase and proton pumps has been obtained. In this study, cryo-electron tomography was used to reveal the supramolecular architecture of the rat heart mitochondria cristae during ATP synthesis. Respirasome and ATP synthase structure in situ were determined using subtomogram averaging. The obtained reconstructions of the inner mitochondrial membrane demonstrated that rows of respiratory chain supercomplexes can dock with rows of ATP synthases forming oligomeric ordered clusters. These ordered clusters indicate a new type of OXPHOS structural organization. It should ensure the quickness, efficiency, and damage resistance of OXPHOS, providing a direct proton transfer from pumps to ATP synthase along the lateral pH gradient without energy dissipation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11605.map.gz emd_11605.map.gz | 782.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11605-v30.xml emd-11605-v30.xml emd-11605.xml emd-11605.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11605_fsc.xml emd_11605_fsc.xml | 2.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11605.png emd_11605.png | 76.2 KB | ||
| マスクデータ |  emd_11605_msk_1.map emd_11605_msk_1.map | 844.8 KB |  マスクマップ マスクマップ | |
| その他 |  emd_11605_half_map_1.map.gz emd_11605_half_map_1.map.gz emd_11605_half_map_2.map.gz emd_11605_half_map_2.map.gz | 607.8 KB 605.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11605 http://ftp.pdbj.org/pub/emdb/structures/EMD-11605 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11605 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11605 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11605.map.gz / 形式: CCP4 / 大きさ: 844.7 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11605.map.gz / 形式: CCP4 / 大きさ: 844.7 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 7.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11605_msk_1.map emd_11605_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
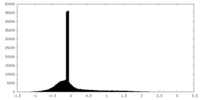
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11605_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
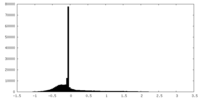
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11605_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Respiratory chain supercomplex I1III2IV1 in rat heart mitochondria
| 全体 | 名称: Respiratory chain supercomplex I1III2IV1 in rat heart mitochondria |
|---|---|
| 要素 |
|
-超分子 #1: Respiratory chain supercomplex I1III2IV1 in rat heart mitochondria
| 超分子 | 名称: Respiratory chain supercomplex I1III2IV1 in rat heart mitochondria タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) / 株: Wistar rats / 器官: heart / Organelle: Mitochondrion Rattus norvegicus (ドブネズミ) / 株: Wistar rats / 器官: heart / Organelle: Mitochondrion |
| 分子量 | 理論値: 1.57 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 2.5 seconds. | ||||||||||
| 詳細 | Dense mitochondrial suspension was diluted to a concentration of 0.3mg/ml in a respiration medium (KCl 80mM, MgCl2 1mM, HEPES/KOH 20mM, KH2PO4 10mM, pH 7.4) and stored for 1 hour without exogenous substrates in a closed microtube on ice (about 4C). Then mitochondria were slowly heated to 20C. 10 minutes prior to vitrification phosphorylation was started by adding 10mM glutamate, 4mM malate, 2mM ADP. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 8.0 µm / 最小 デフォーカス(補正後): 6.0 µm / 倍率(補正後): 37837 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 6.0 µm / 倍率(公称値): 18000 Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 6.0 µm / 倍率(公称値): 18000 |
| 特殊光学系 | 球面収差補正装置: Cs image corrector (CEOS, Germany) |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 平均露光時間: 1.5 sec. / 平均電子線量: 1.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X