+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1093 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM visualization of a viral internal ribosome entry site bound to human ribosomes: the IRES functions as an RNA-based translation factor. | |||||||||
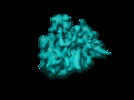
 マップデータ マップデータ | map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 18.3 Å クライオ電子顕微鏡法 / 解像度: 18.3 Å | |||||||||
 データ登録者 データ登録者 | Spahn CM / Frank J | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2004 ジャーナル: Cell / 年: 2004タイトル: Cryo-EM visualization of a viral internal ribosome entry site bound to human ribosomes: the IRES functions as an RNA-based translation factor. 著者: Christian M T Spahn / Eric Jan / Anke Mulder / Robert A Grassucci / Peter Sarnow / Joachim Frank /  要旨: Internal initiation of protein synthesis in eukaryotes is accomplished by recruitment of ribosomes to structured internal ribosome entry sites (IRESs), which are located in certain viral and cellular ...Internal initiation of protein synthesis in eukaryotes is accomplished by recruitment of ribosomes to structured internal ribosome entry sites (IRESs), which are located in certain viral and cellular messenger RNAs. An IRES element in cricket paralysis virus (CrPV) can directly assemble 80S ribosomes in the absence of canonical initiation factors and initiator tRNA. Here we present cryo-EM structures of the CrPV IRES bound to the human ribosomal 40S subunit and to the 80S ribosome. The CrPV IRES adopts a defined, elongate structure within the ribosomal intersubunit space and forms specific contacts with components of the ribosomal A, P, and E sites. Conformational changes in the ribosome as well as within the IRES itself show that CrPV IRES actively manipulates the ribosome. CrPV-like IRES elements seem to act as RNA-based translation factors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1093.map.gz emd_1093.map.gz | 7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1093-v30.xml emd-1093-v30.xml emd-1093.xml emd-1093.xml | 7.5 KB 7.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1093.gif 1093.gif | 43.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1093 http://ftp.pdbj.org/pub/emdb/structures/EMD-1093 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1093 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1093 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1093.map.gz / 形式: CCP4 / 大きさ: 7.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1093.map.gz / 形式: CCP4 / 大きさ: 7.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.66 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human ribosome 80s
| 全体 | 名称: Human ribosome 80s |
|---|---|
| 要素 |
|
-超分子 #1000: Human ribosome 80s
| 超分子 | 名称: Human ribosome 80s / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Human ribosome 80s
| 超分子 | 名称: Human ribosome 80s / タイプ: complex / ID: 1 / 組換発現: No / Ribosome-details: ribosome-eukaryote: ALL |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 凍結 | 凍結剤: ETHANE |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 38000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.26 mm / 倍率(公称値): 39000 Bright-field microscopy / Cs: 2.26 mm / 倍率(公称値): 39000 |
| 試料ステージ | 試料ホルダー: FEI Polara cartridge system / 試料ホルダーモデル: OTHER |
| 温度 | 平均: 84 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 100,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 14 µm / 平均電子線量: 20 e/Å2 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: defocus group volumes |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 18.3 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER |
 ムービー
ムービー コントローラー
コントローラー