+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10886 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HSPD1 single ring prepared by blotting | ||||||||||||
 マップデータ マップデータ | unsharpened final map | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報coated vesicle / isotype switching to IgG isotypes /  mitochondrial unfolded protein response / lipopolysaccharide receptor complex / mitochondrial unfolded protein response / lipopolysaccharide receptor complex /  apolipoprotein A-I binding / TFAP2A acts as a transcriptional repressor during retinoic acid induced cell differentiation / protein import into mitochondrial intermembrane space / migrasome / high-density lipoprotein particle binding / positive regulation of T cell mediated immune response to tumor cell ...coated vesicle / isotype switching to IgG isotypes / apolipoprotein A-I binding / TFAP2A acts as a transcriptional repressor during retinoic acid induced cell differentiation / protein import into mitochondrial intermembrane space / migrasome / high-density lipoprotein particle binding / positive regulation of T cell mediated immune response to tumor cell ...coated vesicle / isotype switching to IgG isotypes /  mitochondrial unfolded protein response / lipopolysaccharide receptor complex / mitochondrial unfolded protein response / lipopolysaccharide receptor complex /  apolipoprotein A-I binding / TFAP2A acts as a transcriptional repressor during retinoic acid induced cell differentiation / protein import into mitochondrial intermembrane space / migrasome / high-density lipoprotein particle binding / positive regulation of T cell mediated immune response to tumor cell / apolipoprotein A-I binding / TFAP2A acts as a transcriptional repressor during retinoic acid induced cell differentiation / protein import into mitochondrial intermembrane space / migrasome / high-density lipoprotein particle binding / positive regulation of T cell mediated immune response to tumor cell /  chaperonin ATPase / chaperonin ATPase /  Mitochondrial protein import / positive regulation of macrophage activation / cellular response to interleukin-7 / biological process involved in interaction with symbiont / MyD88-dependent toll-like receptor signaling pathway / 'de novo' protein folding / sperm plasma membrane / B cell activation / Mitochondrial protein import / positive regulation of macrophage activation / cellular response to interleukin-7 / biological process involved in interaction with symbiont / MyD88-dependent toll-like receptor signaling pathway / 'de novo' protein folding / sperm plasma membrane / B cell activation /  DNA replication origin binding / B cell proliferation / apoptotic mitochondrial changes / positive regulation of interferon-alpha production / DNA replication origin binding / B cell proliferation / apoptotic mitochondrial changes / positive regulation of interferon-alpha production /  apolipoprotein binding / positive regulation of interleukin-10 production / protein maturation / response to unfolded protein / chaperone-mediated protein complex assembly / apolipoprotein binding / positive regulation of interleukin-10 production / protein maturation / response to unfolded protein / chaperone-mediated protein complex assembly /  クラスリン / クラスリン /  isomerase activity / sperm midpiece / response to cold / positive regulation of interleukin-12 production / isomerase activity / sperm midpiece / response to cold / positive regulation of interleukin-12 production /  T細胞 / T細胞 /  分泌 / 分泌 /  lipopolysaccharide binding / ATP-dependent protein folding chaperone / positive regulation of interleukin-6 production / activation of cysteine-type endopeptidase activity involved in apoptotic process / positive regulation of T cell activation / unfolded protein binding / positive regulation of type II interferon production / lipopolysaccharide binding / ATP-dependent protein folding chaperone / positive regulation of interleukin-6 production / activation of cysteine-type endopeptidase activity involved in apoptotic process / positive regulation of T cell activation / unfolded protein binding / positive regulation of type II interferon production /  p53 binding / p53 binding /  フォールディング / フォールディング /  single-stranded DNA binding / single-stranded DNA binding /  double-stranded RNA binding / protein-folding chaperone binding / protein refolding / double-stranded RNA binding / protein-folding chaperone binding / protein refolding /  ミトコンドリア内膜 / protein stabilization / ミトコンドリア内膜 / protein stabilization /  エンドソーム / エンドソーム /  ミトコンドリアマトリックス / positive regulation of apoptotic process / ミトコンドリアマトリックス / positive regulation of apoptotic process /  ubiquitin protein ligase binding / negative regulation of apoptotic process / ubiquitin protein ligase binding / negative regulation of apoptotic process /  enzyme binding / enzyme binding /  細胞膜 / 細胞膜 /  ATP hydrolysis activity / protein-containing complex / ATP hydrolysis activity / protein-containing complex /  ミトコンドリア / ミトコンドリア /  extracellular space / extracellular space /  RNA binding / extracellular exosome / RNA binding / extracellular exosome /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
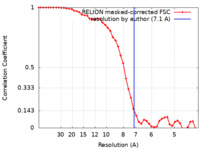
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.1 Å クライオ電子顕微鏡法 / 解像度: 7.1 Å | ||||||||||||
 データ登録者 データ登録者 | Klebl DP / Gravett MSC / Darrow M / Thompson RF / Muench SP | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2020 ジャーナル: Structure / 年: 2020タイトル: Need for Speed: Examining Protein Behavior during CryoEM Grid Preparation at Different Timescales. 著者: David P Klebl / Molly S C Gravett / Dimitrios Kontziampasis / David J Wright / Robin S Bon / Diana C F Monteiro / Martin Trebbin / Frank Sobott / Howard D White / Michele C Darrow / Rebecca F ...著者: David P Klebl / Molly S C Gravett / Dimitrios Kontziampasis / David J Wright / Robin S Bon / Diana C F Monteiro / Martin Trebbin / Frank Sobott / Howard D White / Michele C Darrow / Rebecca F Thompson / Stephen P Muench /    要旨: A host of new technologies are under development to improve the quality and reproducibility of cryoelectron microscopy (cryoEM) grid preparation. Here we have systematically investigated the ...A host of new technologies are under development to improve the quality and reproducibility of cryoelectron microscopy (cryoEM) grid preparation. Here we have systematically investigated the preparation of three macromolecular complexes using three different vitrification devices (Vitrobot, chameleon, and a time-resolved cryoEM device) on various timescales, including grids made within 6 ms (the fastest reported to date), to interrogate particle behavior at the air-water interface for different timepoints. Results demonstrate that different macromolecular complexes can respond to the thin-film environment formed during cryoEM sample preparation in highly variable ways, shedding light on why cryoEM sample preparation can be difficult to optimize. We demonstrate that reducing time between sample application and vitrification is just one tool to improve cryoEM grid quality, but that it is unlikely to be a generic "silver bullet" for improving the quality of every cryoEM sample preparation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10886.map.gz emd_10886.map.gz | 6.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10886-v30.xml emd-10886-v30.xml emd-10886.xml emd-10886.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10886_fsc.xml emd_10886_fsc.xml | 4.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10886.png emd_10886.png | 76.5 KB | ||
| マスクデータ |  emd_10886_msk_1.map emd_10886_msk_1.map | 7.3 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10886_half_map_1.map.gz emd_10886_half_map_1.map.gz emd_10886_half_map_2.map.gz emd_10886_half_map_2.map.gz | 5.5 MB 5.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10886 http://ftp.pdbj.org/pub/emdb/structures/EMD-10886 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10886 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10886 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10886.map.gz / 形式: CCP4 / 大きさ: 7.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10886.map.gz / 形式: CCP4 / 大きさ: 7.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened final map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.13 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10886_msk_1.map emd_10886_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10886_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10886_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human mitochondrial 60 kDa heat shock protein (mature)
| 全体 | 名称: human mitochondrial 60 kDa heat shock protein (mature) |
|---|---|
| 要素 |
|
-超分子 #1: human mitochondrial 60 kDa heat shock protein (mature)
| 超分子 | 名称: human mitochondrial 60 kDa heat shock protein (mature) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: human mitochondrial 60 kDa heat shock protein (mature)
| 分子 | 名称: human mitochondrial 60 kDa heat shock protein (mature) タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SNAAKDVKFG ADARALMLQG VDLLADAVAV TMGPKGRTVI IEQSWGSPKV TKDGVTVAKS IDLKDKYKNI GAKLVQDVAN NTNEEAGDGT TTATVLARSI AKEGFEKISK GANPVEIRRG VMLAVDAVIA ELKKQSKPVT TPEEIAQVAT ISANGDKEIG NIISDAMKKV ...文字列: SNAAKDVKFG ADARALMLQG VDLLADAVAV TMGPKGRTVI IEQSWGSPKV TKDGVTVAKS IDLKDKYKNI GAKLVQDVAN NTNEEAGDGT TTATVLARSI AKEGFEKISK GANPVEIRRG VMLAVDAVIA ELKKQSKPVT TPEEIAQVAT ISANGDKEIG NIISDAMKKV GRKGVITVKD GKTLNDELEI IEGMKFDRGY ISPYFINTSK GQKCEFQDAY VLLSEKKISS IQSIVPALEI ANAHRKPLVI IAEDVDGEAL STLVLNRLKV GLQVVAVKAP GFGDNRKNQL KDMAIATGGA VFGEEGLTLN LEDVQPHDLG KVGEVIVTKD DAMLLKGKGD KAQIEKRIQE IIEQLDVTTS EYEKEKLNER LAKLSDGVAV LKVGGTSDVE VNEKKDRVTD ALNATRAAVE EGIVLGGGCA LLRCIPALDS LTPANEDQKI GIEIIKRTLK IPAMTIAKNA GVEGSLIVEK IMQSSSEVGY DAMAGDFVNM VEKGIIDPTK VVRTALLDAA GVASLLTTAE VVVTEIPKEE KDPGMGAMGG MGGGMGGGMF |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.23 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV / 詳細: blot time 6 s blot force 6. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均露光時間: 1.5 sec. / 平均電子線量: 75.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X