[日本語] English
 万見
万見- EMDB-10460: Sub-tomogram average of the Sulfolobus acidocaldarius S-layer in situ -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10460 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
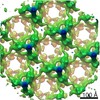
| タイトル | Sub-tomogram average of the Sulfolobus acidocaldarius S-layer in situ | |||||||||
 マップデータ マップデータ | Sub-tomogram averaging of the Sulfolobus acidocaldarius S-layer. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |    Sulfolobus islandicus (好気性・好酸性) Sulfolobus islandicus (好気性・好酸性) | |||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 56.0 Å クライオ電子顕微鏡法 / 解像度: 56.0 Å | |||||||||
 データ登録者 データ登録者 | Daum B / Gambelli L | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2019 ジャーナル: Proc Natl Acad Sci U S A / 年: 2019タイトル: Architecture and modular assembly of S-layers revealed by electron cryotomography. 著者: Lavinia Gambelli / Benjamin H Meyer / Mathew McLaren / Kelly Sanders / Tessa E F Quax / Vicki A M Gold / Sonja-Verena Albers / Bertram Daum /   要旨: Surface protein layers (S-layers) often form the only structural component of the archaeal cell wall and are therefore important for cell survival. S-layers have a plethora of cellular functions ...Surface protein layers (S-layers) often form the only structural component of the archaeal cell wall and are therefore important for cell survival. S-layers have a plethora of cellular functions including maintenance of cell shape, osmotic, and mechanical stability, the formation of a semipermeable protective barrier around the cell, and cell-cell interaction, as well as surface adhesion. Despite the central importance of S-layers for archaeal life, their 3-dimensional (3D) architecture is still poorly understood. Here we present detailed 3D electron cryomicroscopy maps of archaeal S-layers from 3 different strains. We were able to pinpoint the positions and determine the structure of the 2 subunits SlaA and SlaB. We also present a model describing the assembly of the mature S-layer. #1:  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Architecture and modular assembly of Sulfolobus S-layers revealed by electron cryo-tomography 著者: Gambelli L / Meyer BH / McLaren M / Sanders K / Quax TEF / Gold VAM / Albers SVA / Daum B | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10460.map.gz emd_10460.map.gz | 984.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10460-v30.xml emd-10460-v30.xml emd-10460.xml emd-10460.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10460.png emd_10460.png | 112.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10460 http://ftp.pdbj.org/pub/emdb/structures/EMD-10460 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10460 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10460 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10460.map.gz / 形式: CCP4 / 大きさ: 1.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10460.map.gz / 形式: CCP4 / 大きさ: 1.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-tomogram averaging of the Sulfolobus acidocaldarius S-layer. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 10.73 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SlaA/SlaB array
| 全体 | 名称: SlaA/SlaB array |
|---|---|
| 要素 |
|
-超分子 #1: SlaA/SlaB array
| 超分子 | 名称: SlaA/SlaB array / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:    Sulfolobus islandicus (好気性・好酸性) Sulfolobus islandicus (好気性・好酸性) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 3 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | エネルギーフィルター - スリット幅: 40 eV |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) #0 - 検出モード: COUNTING / #0 - 平均電子線量: 1.8 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) #1 - 平均電子線量: 1.8 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 抽出 | トモグラム数: 1 / 使用した粒子像数: 2068 |
|---|---|
| CTF補正 | ソフトウェア - 名称:  IMOD IMOD |
| 最終 角度割当 | タイプ: OTHER / ソフトウェア - 名称: PEET |
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 56.0 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES / ソフトウェア - 名称: PEET / 使用したサブトモグラム数: 2068 ) / 解像度のタイプ: BY AUTHOR / 解像度: 56.0 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES / ソフトウェア - 名称: PEET / 使用したサブトモグラム数: 2068 |
| Image recording ID | 1 |
 ムービー
ムービー コントローラー
コントローラー