+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10160 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In Situ Core-Signalling Unit of E. coli Chemoreceptor Array | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 16.0 Å クライオ電子顕微鏡法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Burt A / Desfosses A / Gutsche I | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Complete structure of the chemosensory array core signalling unit in an E. coli minicell strain. 著者: Alister Burt / C Keith Cassidy / Peter Ames / Maria Bacia-Verloop / Megghane Baulard / Karine Huard / Zaida Luthey-Schulten / Ambroise Desfosses / Phillip J Stansfeld / William Margolin / ...著者: Alister Burt / C Keith Cassidy / Peter Ames / Maria Bacia-Verloop / Megghane Baulard / Karine Huard / Zaida Luthey-Schulten / Ambroise Desfosses / Phillip J Stansfeld / William Margolin / John S Parkinson / Irina Gutsche /    要旨: Motile bacteria sense chemical gradients with transmembrane receptors organised in supramolecular signalling arrays. Understanding stimulus detection and transmission at the molecular level requires ...Motile bacteria sense chemical gradients with transmembrane receptors organised in supramolecular signalling arrays. Understanding stimulus detection and transmission at the molecular level requires precise structural characterisation of the array building block known as a core signalling unit. Here we introduce an Escherichia coli strain that forms small minicells possessing extended and highly ordered chemosensory arrays. We use cryo-electron tomography and subtomogram averaging to provide a three-dimensional map of a complete core signalling unit, with visible densities corresponding to the HAMP and periplasmic domains. This map, combined with previously determined high resolution structures and molecular dynamics simulations, yields a molecular model of the transmembrane core signalling unit and enables spatial localisation of its individual domains. Our work thus offers a solid structural basis for the interpretation of a wide range of existing data and the design of further experiments to elucidate signalling mechanisms within the core signalling unit and larger array. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10160.map.gz emd_10160.map.gz | 4.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10160-v30.xml emd-10160-v30.xml emd-10160.xml emd-10160.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10160_fsc.xml emd_10160_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10160.png emd_10160.png | 55.5 KB | ||
| マスクデータ |  emd_10160_msk_1.map emd_10160_msk_1.map emd_10160_msk_2.map emd_10160_msk_2.map | 8 MB 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10160_additional.map.gz emd_10160_additional.map.gz emd_10160_additional_1.map.gz emd_10160_additional_1.map.gz emd_10160_half_map_1.map.gz emd_10160_half_map_1.map.gz emd_10160_half_map_2.map.gz emd_10160_half_map_2.map.gz | 4.5 MB 4.5 MB 7.4 MB 7.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10160 http://ftp.pdbj.org/pub/emdb/structures/EMD-10160 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10160 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10160 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10364 (タイトル: Cryo-electron tomography of E. coli minicells / Data size: 141.7 EMPIAR-10364 (タイトル: Cryo-electron tomography of E. coli minicells / Data size: 141.7 Data #1: Unaligned multi-frame micrographs from tilt series containing WM4196 minicells [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10160.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10160.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.48 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10160_msk_1.map emd_10160_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
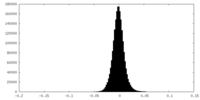
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_10160_msk_2.map emd_10160_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
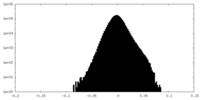
| 密度ヒストグラム |
-追加マップ: Local resolution filtered map centered on 3fold axis...
| ファイル | emd_10160_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution filtered map centered on 3fold axis of chemoreceptor array. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
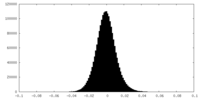
| 密度ヒストグラム |
-追加マップ: Local resolution filtered map centered on 3fold axis...
| ファイル | emd_10160_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution filtered map centered on 3fold axis of chemoreceptor array. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: even half map.
| ファイル | emd_10160_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | even half map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: odd half map.
| ファイル | emd_10160_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | odd half map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Optimised E. coli minicell strain WM4196 containing native-state ...
| 全体 | 名称: Optimised E. coli minicell strain WM4196 containing native-state chemoreceptor arrays. |
|---|---|
| 要素 |
|
-超分子 #1: Optimised E. coli minicell strain WM4196 containing native-state ...
| 超分子 | 名称: Optimised E. coli minicell strain WM4196 containing native-state chemoreceptor arrays. タイプ: cell / ID: 1 / 親要素: 0 詳細: Minicells isolated from WM4196 cell culture by differential centrifugation. |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: WM4196 Escherichia coli (大腸菌) / 株: WM4196 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R3.5/1 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: 25 mA |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最小 デフォーカス(公称値): 0.0002 µm Bright-field microscopy / 最小 デフォーカス(公称値): 0.0002 µm |
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-5 / 撮影したグリッド数: 1 / 平均電子線量: 1.0 e/Å2 詳細: Images were collected in movie mode, 5 frames per movie. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X