+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7 | |||||||||||||||||||||||||||
 マップデータ マップデータ | SARS-CoV-2 spike (S) protein trimer in complex with anti-receptor binding domain (RBD) Fab E7 | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) VIRAL PROTEIN (ウイルスタンパク質) | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell /  receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||||||||
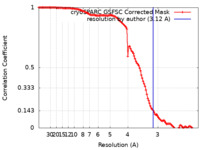
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.12 Å クライオ電子顕微鏡法 / 解像度: 3.12 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Chia WN / Tan CW / Tan AWK / Young B / Starr TN / Lopez E / Fibriansah G / Barr J / Cheng S / Yeoh AYY ...Chia WN / Tan CW / Tan AWK / Young B / Starr TN / Lopez E / Fibriansah G / Barr J / Cheng S / Yeoh AYY / Yap WC / Lim BL / Ng TS / Sia WR / Zhu F / Chen S / Zhang J / Greaney AJ / Chen M / Au GG / Paradkar P / Peiris M / Chung AW / Bloom JD / Lye D / Lok SM / Wang LF | |||||||||||||||||||||||||||
| 資金援助 |  シンガポール, シンガポール,  オーストラリア, オーストラリア,  米国, 8件 米国, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Potent pan huACE2-dependent sarbecovirus neutralizing monoclonal antibodies isolated from a BNT162b2-vaccinated SARS survivor. 著者: Wan Ni Chia / Chee Wah Tan / Aaron Wai Kit Tan / Barnaby Young / Tyler N Starr / Ester Lopez / Guntur Fibriansah / Jennifer Barr / Samuel Cheng / Aileen Ying-Yan Yeoh / Wee Chee Yap / Beng ...著者: Wan Ni Chia / Chee Wah Tan / Aaron Wai Kit Tan / Barnaby Young / Tyler N Starr / Ester Lopez / Guntur Fibriansah / Jennifer Barr / Samuel Cheng / Aileen Ying-Yan Yeoh / Wee Chee Yap / Beng Lee Lim / Thiam-Seng Ng / Wan Rong Sia / Feng Zhu / Shiwei Chen / Jinyan Zhang / Madeline Sheng Si Kwek / Allison J Greaney / Mark Chen / Gough G Au / Prasad N Paradkar / Malik Peiris / Amy W Chung / Jesse D Bloom / David Lye / Sheemei Lok / Lin-Fa Wang /     要旨: The emergence of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) variants of concern such as Omicron hampered efforts in controlling the ongoing coronavirus disease 2019 pandemic due to ...The emergence of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) variants of concern such as Omicron hampered efforts in controlling the ongoing coronavirus disease 2019 pandemic due to their ability to escape neutralizing antibodies induced by vaccination or prior infection, highlighting the need to develop broad-spectrum vaccines and therapeutics. Most human monoclonal antibodies (mAbs) reported to date have not demonstrated true pan-sarbecovirus neutralizing breadth especially against animal sarbecoviruses. Here, we report the isolation and characterization of highly potent mAbs targeting the receptor binding domain (RBD) of huACE2-dependent sarbecovirus from a SARS-CoV survivor vaccinated with BNT162b2. Among the six mAbs identified, one (E7) showed better huACE2-dependent sarbecovirus neutralizing potency and breadth than any other mAbs reported to date. Mutagenesis and cryo-electron microscopy studies indicate that these mAbs have a unique RBD contact footprint and that E7 binds to a quaternary structure-dependent epitope. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33650.map.gz emd_33650.map.gz | 161.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33650-v30.xml emd-33650-v30.xml emd-33650.xml emd-33650.xml | 25 KB 25 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33650_fsc.xml emd_33650_fsc.xml | 11.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33650.png emd_33650.png | 138.8 KB | ||
| その他 |  emd_33650_half_map_1.map.gz emd_33650_half_map_1.map.gz emd_33650_half_map_2.map.gz emd_33650_half_map_2.map.gz | 165.4 MB 165.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33650 http://ftp.pdbj.org/pub/emdb/structures/EMD-33650 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33650 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33650 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7y71MC  7y72C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33650.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33650.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 spike (S) protein trimer in complex with anti-receptor binding domain (RBD) Fab E7 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.105 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33650_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
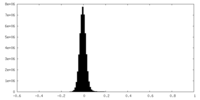
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33650_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
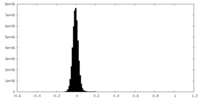
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment ...
| 全体 | 名称: SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment ...
| 超分子 | 名称: SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Fab E7 light chain
| 分子 | 名称: Fab E7 light chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.920668 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIVMTQSPLS LPVTPGEPAS ISCRSSQSLL QNNGYNYLAW YLQKPGQSPQ LLIYLSSTRA SGVPDRFSGS GSGTDFTLKI SRVEAEDVG VYYCMQSLQI PGTFGQGTRL EIKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV ...文字列: DIVMTQSPLS LPVTPGEPAS ISCRSSQSLL QNNGYNYLAW YLQKPGQSPQ LLIYLSSTRA SGVPDRFSGS GSGTDFTLKI SRVEAEDVG VYYCMQSLQI PGTFGQGTRL EIKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV TEQDSKDSTY SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE C |
-分子 #2: Fab E7 heavy chain
| 分子 | 名称: Fab E7 heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.339156 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSETLSL TCTVSGGFIG PHYWSWVRQP PGKGLEWIGY IYISGSTNYN PSLKSRLTIS VDMSKSQFSL TLSSATAAD TAVYYCARGG GYLETGPFEY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS ...文字列: QVQLQESGPG LVKPSETLSL TCTVSGGFIG PHYWSWVRQP PGKGLEWIGY IYISGSTNYN PSLKSRLTIS VDMSKSQFSL TLSSATAAD TAVYYCARGG GYLETGPFEY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKRV EPK |
-分子 #3: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 132.860156 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VNLTTRTQLP PAYTNSFTRG VYYPDKVFRS SVLHSTQDLF LPFFSNVTWF HAIHVSGTNG TKRFDNPVLP FNDGVYFAST EKSNIIRGW IFGTTLDSKT QSLLIVNNAT NVVIKVCEFQ FCNDPFLGVY YHKNNKSWME SEFRVYSSAN NCTFEYVSQP F LMDLEGKQ ...文字列: VNLTTRTQLP PAYTNSFTRG VYYPDKVFRS SVLHSTQDLF LPFFSNVTWF HAIHVSGTNG TKRFDNPVLP FNDGVYFAST EKSNIIRGW IFGTTLDSKT QSLLIVNNAT NVVIKVCEFQ FCNDPFLGVY YHKNNKSWME SEFRVYSSAN NCTFEYVSQP F LMDLEGKQ GNFKNLREFV FKNIDGYFKI YSKHTPINLV RDLPQGFSAL EPLVDLPIGI NITRFQTLLA LHRSYLTPGD SS SGWTAGA AAYYVGYLQP RTFLLKYNEN GTITDAVDCA LDPLSETKCT LKSFTVEKGI YQTSNFRVQP TESIVRFPNI TNL CPFGEV FNATRFASVY AWNRKRISNC VADYSVLYNS ASFSTFKCYG VSPTKLNDLC FTNVYADSFV IRGDEVRQIA PGQT GKIAD YNYKLPDDFT GCVIAWNSNN LDSKVGGNYN YLYRLFRKSN LKPFERDIST EIYQAGSTPC NGVEGFNCYF PLQSY GFQP TNGVGYQPYR VVVLSFELLH APATVCGPKK STNLVKNKCV NFNFNGLTGT GVLTESNKKF LPFQQFGRDI ADTTDA VRD PQTLEILDIT PCSFGGVSVI TPGTNTSNQV AVLYQDVNCT EVPVAIHADQ LTPTWRVYST GSNVFQTRAG CLIGAEH VN NSYECDIPIG AGICASYQTQ TNSPRAAASV ASQSIIAYTM SLGAENSVAY SNNSIAIPTN FTISVTTEIL PVSMTKTS V DCTMYICGDS TECSNLLLQY GSFCTQLNRA LTGIAVEQDK NTQEVFAQVK QIYKTPPIKD FGGFNFSQIL PDPSKPSKR SPIEDLLFNK VTLADAGFIK QYGDCLGDIA ARDLICAQKF NGLTVLPPLL TDEMIAQYTS ALLAGTITSG WTFGAGPALQ IPFPMQMAY RFNGIGVTQN VLYENQKLIA NQFNSAIGKI QDSLSSTPSA LGKLQDVVNQ NAQALNTLVK QLSSNFGAIS S VLNDILSR LDPPEAEVQI DRLITGRLQS LQTYVTQQLI RAAEIRASAN LAATKMSECV LGQSKRVDFC GKGYHLMSFP QS APHGVVF LHVTYVPAQE KNFTTAPAIC HDGKAHFPRE GVFVSNGTHW FVTQRNFYEP QIITTDNTFV SGNCDVVIGI VNN TVYDPL QPELDSFKEE LDKYFKNHTS PDVDLGDISG INASVVNIQK EIDRLNEVAK NLNESLIDLQ ELGKYEQYIK WP UniProtKB:  スパイクタンパク質 スパイクタンパク質 |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 14 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均露光時間: 4.76 sec. / 平均電子線量: 31.6 e/Å2 詳細: Images were collected in counting mode at 25 frames per movie with an exposure time of 4.76 s per movie. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X