+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6327 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3D reconstruction from the 188 most curved out of 450 fibrils of Abeta(1-40) | |||||||||
 マップデータ マップデータ | 3D reconstruction of Abeta(1-40) fibril before any filtering, symmetrization, or masking. Figure 5A of the primary citation was generated from this map by application of helical symmetry and a B factor of -600A^2, with low-pass filtering to 7.5A. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
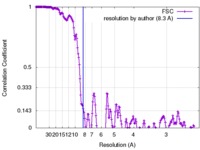
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.3 Å クライオ電子顕微鏡法 / 解像度: 8.3 Å | |||||||||
 データ登録者 データ登録者 | Rohou A / Grigorieff N | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2008 ジャーナル: Proc Natl Acad Sci U S A / 年: 2008タイトル: Paired beta-sheet structure of an Abeta(1-40) amyloid fibril revealed by electron microscopy. 著者: Carsten Sachse / Marcus Fändrich / Nikolaus Grigorieff /  要旨: Alzheimer's disease is a neurodegenerative disorder that is characterized by the cerebral deposition of amyloid fibrils formed by Abeta peptide. Despite their prevalence in Alzheimer's and other ...Alzheimer's disease is a neurodegenerative disorder that is characterized by the cerebral deposition of amyloid fibrils formed by Abeta peptide. Despite their prevalence in Alzheimer's and other neurodegenerative diseases, important details of the structure of amyloid fibrils remain unknown. Here, we present a three-dimensional structure of a mature amyloid fibril formed by Abeta(1-40) peptide, determined by electron cryomicroscopy at approximately 8-A resolution. The fibril consists of two protofilaments, each containing approximately 5-nm-long regions of beta-sheet structure. A local twofold symmetry within each region suggests that pairs of beta-sheets are formed from equivalent parts of two Abeta(1-40) peptides contained in each protofilament. The pairing occurs via tightly packed interfaces, reminiscent of recently reported steric zipper structures. However, unlike these previous structures, the beta-sheet pairing is observed within an amyloid fibril and includes significantly longer amino acid sequences. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6327.map.gz emd_6327.map.gz | 365.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6327-v30.xml emd-6327-v30.xml emd-6327.xml emd-6327.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6327_fsc.xml emd_6327_fsc.xml | 21.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6327.jpg emd_6327.jpg | 10.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6327 http://ftp.pdbj.org/pub/emdb/structures/EMD-6327 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6327 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6327 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6327.map.gz / 形式: CCP4 / 大きさ: 494.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6327.map.gz / 形式: CCP4 / 大きさ: 494.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of Abeta(1-40) fibril before any filtering, symmetrization, or masking. Figure 5A of the primary citation was generated from this map by application of helical symmetry and a B factor of -600A^2, with low-pass filtering to 7.5A. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : human Abeta(1-40)
| 全体 | 名称: human Abeta(1-40) |
|---|---|
| 要素 |
|
-超分子 #1000: human Abeta(1-40)
| 超分子 | 名称: human Abeta(1-40) / タイプ: sample / ID: 1000 / 集合状態: Helical assembly / Number unique components: 1 |
|---|
-分子 #1: Amyloid beta peptide (1-40)
| 分子 | 名称: Amyloid beta peptide (1-40) / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Abeta(1-40) / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human Homo sapiens (ヒト) / 別称: human |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 / 詳細: 50 mM borate |
| グリッド | 詳細: Quantifoil R1.2/1.3 Cu 400 mesh grids |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification carried out in cold room at 277 K. / 手法: Blot for 7 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.629 µm / 最小 デフォーカス(公称値): 2.027 µm / 倍率(公称値): 59000 Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.629 µm / 最小 デフォーカス(公称値): 2.027 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 日付 | 2006年2月9日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7.0 µm / 実像数: 63 / 平均電子線量: 35 e/Å2 |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー