ソフトウェア 名称 バージョン 分類 1.9_1692精密化 3.2 データ抽出 データ削減 データスケーリング 位相決定
精密化 解像度 / SU ML / 交差検証法 / σ(F) / 位相誤差 Rfactor 反射数 %反射 Rfree 0.2105 3381 4.61 % Rwork 0.173 - - obs 0.1748 43607 73.72 %
溶媒の処理 減衰半径 / VDWプローブ半径 原子変位パラメータ Biso max 2 / Biso mean 2 / Biso min 2 精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 3178 0 8 276 3462 Biso mean - - 20.78 33.88 - 残基数 - - - - 416
拘束条件 Refine-ID タイプ Dev ideal 数 X-RAY DIFFRACTION f_bond_d0.011 3248 X-RAY DIFFRACTION f_angle_d1.346 4430 X-RAY DIFFRACTION f_chiral_restr0.052 524 X-RAY DIFFRACTION f_plane_restr0.008 568 X-RAY DIFFRACTION f_dihedral_angle_d15.738 1158
LS精密化 シェル Refine-ID / Total num. of bins used
大きな表を表示 (7 x 24) 大きな表を隠す 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Num. reflection all % reflection obs (%)1.5317-1.5536 0.4366 19 0.2908 377 396 10 1.5536-1.5768 0.2916 43 0.3046 889 932 22 1.5768-1.6015 0.2906 63 0.2802 1207 1270 31 1.6015-1.6277 0.3221 75 0.2594 1580 1655 40 1.6277-1.6558 0.3001 102 0.245 1920 2022 48 1.6558-1.6859 0.2639 109 0.2308 2164 2273 56 1.6859-1.7183 0.2664 129 0.2289 2595 2724 64 1.7183-1.7534 0.2623 147 0.2151 3040 3187 78 1.7534-1.7915 0.2016 155 0.2074 3263 3418 82 1.7915-1.8332 0.2388 156 0.2023 3356 3512 84 1.8332-1.879 0.2172 161 0.1936 3349 3510 85 1.879-1.9298 0.2159 171 0.1865 3450 3621 87 1.9298-1.9866 0.2031 160 0.1813 3442 3602 87 1.9866-2.0508 0.2024 170 0.1669 3474 3644 88 2.0508-2.124 0.2053 169 0.1655 3468 3637 88 2.124-2.2091 0.2253 167 0.1728 3478 3645 88 2.2091-2.3096 0.2026 170 0.1682 3503 3673 89 2.3096-2.4314 0.2009 170 0.167 3559 3729 90 2.4314-2.5837 0.2311 168 0.1753 3560 3728 90 2.5837-2.7832 0.203 171 0.1713 3592 3763 90 2.7832-3.0632 0.2415 173 0.1837 3610 3783 92 3.0632-3.5063 0.1843 174 0.1673 3655 3829 92 3.5063-4.4169 0.1907 178 0.1471 3691 3869 93 4.4169-43.9984 0.1996 181 0.1599 3791 3972 96
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 2) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 0.5311 -0.1648 -0.0079 1.1836 0.2749 0.4729 0.0048 -0.026 -0.0398 0.1399 -0.0596 0.0861 0.095 -0.0564 -0.0041 0.151 -0.023 0.0254 0.1281 -0.0081 0.1124 9.2367 -3.634 33.433 2 0.7412 -0.407 -0.0804 1.5411 -0.2423 0.4756 0.1032 0.1454 0.0007 -0.1638 -0.1272 -0.049 0.0307 0.0154 -0.0015 0.1204 0.0272 -0.0142 0.1651 0.0142 0.1092 -0.9905 -8.2027 9.7275
精密化 TLSグループ ID Refine-ID Refine TLS-ID Selection details Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 chain AA1 - 208 2 X-RAY DIFFRACTION 2 chain BB1 - 208
 データを開く
データを開く 基本情報
基本情報 要素
要素
 キーワード
キーワード LYASE (リアーゼ) / METAL BINDING PROTEIN /
LYASE (リアーゼ) / METAL BINDING PROTEIN /  Bacterial Proteins (細菌) /
Bacterial Proteins (細菌) /  Cysteine (システイン) /
Cysteine (システイン) /  Escherichia coli (大腸菌) /
Escherichia coli (大腸菌) /  Lyases (リアーゼ) /
Lyases (リアーゼ) /  Mercury (水銀) / Dimethyltin
Mercury (水銀) / Dimethyltin 機能・相同性情報
機能・相同性情報
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  シンクロトロン / 解像度: 1.532 Å
シンクロトロン / 解像度: 1.532 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J. Am. Chem. Soc. / 年: 2017
ジャーナル: J. Am. Chem. Soc. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5u7a.cif.gz
5u7a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5u7a.ent.gz
pdb5u7a.ent.gz PDB形式
PDB形式 5u7a.json.gz
5u7a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/u7/5u7a
https://data.pdbj.org/pub/pdb/validation_reports/u7/5u7a ftp://data.pdbj.org/pub/pdb/validation_reports/u7/5u7a
ftp://data.pdbj.org/pub/pdb/validation_reports/u7/5u7a リンク
リンク 集合体
集合体
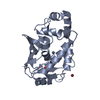

 要素
要素 / Organomercurial lyase
/ Organomercurial lyase
 Escherichia coli (大腸菌) / 遺伝子: merB / 発現宿主:
Escherichia coli (大腸菌) / 遺伝子: merB / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P77072,
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P77072,  alkylmercury lyase
alkylmercury lyase 臭化物
臭化物 リン酸塩
リン酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X25 / 波長: 1.1 Å
/ ビームライン: X25 / 波長: 1.1 Å : 1.1 Å / 相対比: 1
: 1.1 Å / 相対比: 1  解析
解析 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj








