+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3iyd | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional EM structure of an intact activator-dependent transcription initiation complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSCRIPTION/DNA /  transcription (転写 (生物学)) / initiation / Class I / activator / transcription (転写 (生物学)) / initiation / Class I / activator /  RNA polymerase (RNAポリメラーゼ) / RNA polymerase (RNAポリメラーゼ) /  holoenzyme (酵素) / sigma70 / open complex / CAP / CRP / holoenzyme (酵素) / sigma70 / open complex / CAP / CRP /  cAMP-dependent / cAMP-dependent /  DNA (デオキシリボ核酸) / DNA (デオキシリボ核酸) /  prokaryotic (原核生物) / prokaryotic (原核生物) /  DNA-directed RNA polymerase (ポリメラーゼ) / DNA-directed RNA polymerase (ポリメラーゼ) /  Nucleotidyltransferase / Nucleotidyltransferase /  Transferase (転移酵素) / DNA-binding / Transferase (転移酵素) / DNA-binding /  Sigma factor (シグマ因子) / Sigma factor (シグマ因子) /  Transcription regulation / Transcription regulation /  cAMP (CAMP) / cAMP-binding / Nucleotide-binding / TRANSCRIPTION-DNA COMPLEX cAMP (CAMP) / cAMP-binding / Nucleotide-binding / TRANSCRIPTION-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報carbon catabolite repression of transcription / sigma factor antagonist complex / DNA binding, bending /  RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /  sigma factor activity ...carbon catabolite repression of transcription / sigma factor antagonist complex / DNA binding, bending / sigma factor activity ...carbon catabolite repression of transcription / sigma factor antagonist complex / DNA binding, bending /  RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /  sigma factor activity / minor groove of adenine-thymine-rich DNA binding / bacterial-type flagellum-dependent cell motility / nitrate assimilation / sigma factor activity / minor groove of adenine-thymine-rich DNA binding / bacterial-type flagellum-dependent cell motility / nitrate assimilation /  cAMP binding / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination / cAMP binding / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination /  運動性 / 運動性 /  デオキシリボ核酸 / DNA-templated transcription initiation / デオキシリボ核酸 / DNA-templated transcription initiation /  ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity /  ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis / sequence-specific DNA binding / ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis / sequence-specific DNA binding /  protein dimerization activity / DNA-binding transcription factor activity / response to antibiotic / negative regulation of DNA-templated transcription / DNA-templated transcription / positive regulation of DNA-templated transcription / magnesium ion binding / protein dimerization activity / DNA-binding transcription factor activity / response to antibiotic / negative regulation of DNA-templated transcription / DNA-templated transcription / positive regulation of DNA-templated transcription / magnesium ion binding /  DNA binding / zinc ion binding / DNA binding / zinc ion binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌)  Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 19.8 Å ネガティブ染色法 / 解像度: 19.8 Å | ||||||
 データ登録者 データ登録者 | Hudson, B.P. / Quispe, J. / Lara, S. / Kim, Y. / Berman, H. / Arnold, E. / Ebright, R.H. / Lawson, C.L. | ||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2009 ジャーナル: Proc Natl Acad Sci U S A / 年: 2009タイトル: Three-dimensional EM structure of an intact activator-dependent transcription initiation complex. 著者: Brian P Hudson / Joel Quispe / Samuel Lara-González / Younggyu Kim / Helen M Berman / Eddy Arnold / Richard H Ebright / Catherine L Lawson /  要旨: We present the experimentally determined 3D structure of an intact activator-dependent transcription initiation complex comprising the Escherichia coli catabolite activator protein (CAP), RNA ...We present the experimentally determined 3D structure of an intact activator-dependent transcription initiation complex comprising the Escherichia coli catabolite activator protein (CAP), RNA polymerase holoenzyme (RNAP), and a DNA fragment containing positions -78 to +20 of a Class I CAP-dependent promoter with a CAP site at position -61.5 and a premelted transcription bubble. A 20-A electron microscopy reconstruction was obtained by iterative projection-based matching of single particles visualized in carbon-sandwich negative stain and was fitted using atomic coordinate sets for CAP, RNAP, and DNA. The structure defines the organization of a Class I CAP-RNAP-promoter complex and supports previously proposed interactions of CAP with RNAP alpha subunit C-terminal domain (alphaCTD), interactions of alphaCTD with sigma(70) region 4, interactions of CAP and RNAP with promoter DNA, and phased-DNA-bend-dependent partial wrapping of DNA around the complex. The structure also reveals the positions and shapes of species-specific domains within the RNAP beta', beta, and sigma(70) subunits. | ||||||
| 履歴 |
| ||||||
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR DETERMINED | ||||||
| Remark 700 | SHEET DETERMINATION METHOD: AUTHOR DETERMINED |
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3iyd.cif.gz 3iyd.cif.gz | 778.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3iyd.ent.gz pdb3iyd.ent.gz | 598.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3iyd.json.gz 3iyd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/iy/3iyd https://data.pdbj.org/pub/pdb/validation_reports/iy/3iyd ftp://data.pdbj.org/pub/pdb/validation_reports/iy/3iyd ftp://data.pdbj.org/pub/pdb/validation_reports/iy/3iyd | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-DNA-directed RNA polymerase subunit ... , 4種, 5分子 ABCDE
| #1: タンパク質 |  ポリメラーゼ / RNAP subunit alpha / Transcriptase subunit alpha / RNA polymerase subunit alpha ポリメラーゼ / RNAP subunit alpha / Transcriptase subunit alpha / RNA polymerase subunit alpha分子量: 36558.680 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌) / 株: K-12 Escherichia coli (大腸菌) / 株: K-12遺伝子: b3295, JW3257, pez, phs, rpoA, RpoB, RpoC, RpoD, RpoZ, sez プラスミド: pEcABC-H6, pRSFduet-sigma, pCDF-omega / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE2) / 参照: UniProt: P0A7Z4, Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE2) / 参照: UniProt: P0A7Z4,  ポリメラーゼ ポリメラーゼ#2: タンパク質 | |  ポリメラーゼ / RNAP subunit beta / Transcriptase subunit beta / RNA polymerase subunit beta ポリメラーゼ / RNAP subunit beta / Transcriptase subunit beta / RNA polymerase subunit beta分子量: 150820.875 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌) / 株: K-12 Escherichia coli (大腸菌) / 株: K-12遺伝子: rpoB, groN, nitB, rif, ron, stl, stv, tabD, b3987, JW3950 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P0A8V2, Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P0A8V2,  ポリメラーゼ ポリメラーゼ#3: タンパク質 | |  ポリメラーゼ / RNAP subunit beta' / Transcriptase subunit beta' / RNA polymerase subunit beta' ポリメラーゼ / RNAP subunit beta' / Transcriptase subunit beta' / RNA polymerase subunit beta'分子量: 156195.625 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: Purchased from IDT and annealed / 参照: UniProt: P0A8T7,  ポリメラーゼ ポリメラーゼ#4: タンパク質 | |  ポリメラーゼ / RNAP omega subunit / Transcriptase subunit omega / RNA polymerase omega subunit ポリメラーゼ / RNAP omega subunit / Transcriptase subunit omega / RNA polymerase omega subunit分子量: 10118.352 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: rpoZ, b3649, JW3624 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: rpoZ, b3649, JW3624 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P0A800, Escherichia coli (大腸菌) / 参照: UniProt: P0A800,  ポリメラーゼ ポリメラーゼ |
|---|
-タンパク質 , 2種, 3分子 FGH
| #5: タンパク質 | 分子量: 70326.234 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: rpoD / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: rpoD / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P00579 Escherichia coli (大腸菌) / 参照: UniProt: P00579 |
|---|---|
| #6: タンパク質 | 分子量: 23541.242 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: crp, cap, csm, b3357, JW5702 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: crp, cap, csm, b3357, JW5702 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P0ACJ8 Escherichia coli (大腸菌) / 参照: UniProt: P0ACJ8 |
-DNA鎖 , 2種, 2分子 IJ
| #7: DNA鎖 | 分子量: 30220.389 Da / 分子数: 1 / 由来タイプ: 合成 |
|---|---|
| #8: DNA鎖 | 分子量: 30097.295 Da / 分子数: 1 / 由来タイプ: 合成 |
-非ポリマー , 1種, 2分子 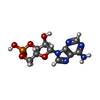
| #9: 化合物 |  環状アデノシン一リン酸 環状アデノシン一リン酸 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.57 MDa / 実験値: NO | |||||||||||||||||||||||||
| 緩衝液 | 名称: 25 mM HEPES, 100 mM KCl, 10 mM MgCl2, 1 mM DTT, 0.2 mM cAMP pH: 8 詳細: 25 mM HEPES, 100 mM KCl, 10 mM MgCl2, 1 mM DTT, 0.2 mM cAMP | |||||||||||||||||||||||||
| 試料 | 濃度: 6.18 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : YES / 凍結 : YES / 凍結 : NO : NO詳細: 25mM HEPES, 100mM KCl, 10mM MgCl2, 1mM DTT, 0.2mM cAMP | |||||||||||||||||||||||||
| 染色 | タイプ: NEGATIVE / 染色剤: Uranyl Formate | |||||||||||||||||||||||||
| 試料支持 | 詳細: 400-mesh copper 2.0x0.5 hole pattern C-flat grids (Protochips, Inc.) with a thin layer of continuous carbon floated on top |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TECNAI F20 / 日付: 2008年11月4日 / 詳細: 15 um pixel size on detector |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 120 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 50000 X / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 500 nm / Cs Bright-field microscopy / 倍率(公称値): 50000 X / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 500 nm / Cs : 2 mm / カメラ長: 0 mm : 2 mm / カメラ長: 0 mm |
| 試料ホルダ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC 資料ホルダタイプ: standard side-entry room-temperature stage 温度: 293 K / 傾斜角・最大: 0 ° / 傾斜角・最小: 0 ° |
| 撮影 | 電子線照射量: 16 e/Å2 フィルム・検出器のモデル: TVIPS TEMCAM-F415 (4k x 4k) |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | 詳細: ACE | ||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||
3次元再構成 | 手法: projection matching / 解像度: 19.8 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 14097 詳細: EMAN interleaved with SPIDER correspondence analysis ( Details about the particle: 32816 particles were automatically selected by the Appion DoGpicker initially ) クラス平均像の数: 280 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL / Target criteria: map-derived potential energy 詳細: REFINEMENT PROTOCOL--rigid body, Yup.scx simulated annealing DETAILS--A complete ternary complex model was generated using multiple PDB entries, with a homology modelling step for RNAP. The ...詳細: REFINEMENT PROTOCOL--rigid body, Yup.scx simulated annealing DETAILS--A complete ternary complex model was generated using multiple PDB entries, with a homology modelling step for RNAP. The model was regularized with PHENIX and the refined against the EM map with Yup.scx using default parameters. | ||||||||||||||||||||||||||||||
| 原子モデル構築 | 3D fitting-ID: 1 / Source name: PDB / タイプ: experimental model
| ||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj














































