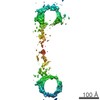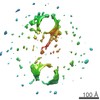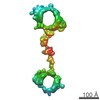+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2948 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three Dimensional Dynamics and Fluctuations of DNA-Nanogold Dimers by Individual-Particle Electron Tomography | |||||||||
 マップデータ マップデータ | Reconstruction of one particle of DNA-nanogold dimer by using individual-particle electron tomography | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | 3D structure / DNA-nanogold conjugate / individual-particle electron tomography / IPET | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 |  電子線トモグラフィー法 / 電子線トモグラフィー法 /  ネガティブ染色法 / 解像度: 14.7 Å ネガティブ染色法 / 解像度: 14.7 Å | |||||||||
 データ登録者 データ登録者 | Zhang L / Smith JM / Tong HM / Zhang X / Lei DS / Lu ZY / Alivisatos P / Ren G | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2016 ジャーナル: Nat Commun / 年: 2016タイトル: Three-dimensional structural dynamics and fluctuations of DNA-nanogold conjugates by individual-particle electron tomography. 著者: Lei Zhang / Dongsheng Lei / Jessica M Smith / Meng Zhang / Huimin Tong / Xing Zhang / Zhuoyang Lu / Jiankang Liu / A Paul Alivisatos / Gang Ren /   要旨: DNA base pairing has been used for many years to direct the arrangement of inorganic nanocrystals into small groupings and arrays with tailored optical and electrical properties. The control of DNA- ...DNA base pairing has been used for many years to direct the arrangement of inorganic nanocrystals into small groupings and arrays with tailored optical and electrical properties. The control of DNA-mediated assembly depends crucially on a better understanding of three-dimensional structure of DNA-nanocrystal-hybridized building blocks. Existing techniques do not allow for structural determination of these flexible and heterogeneous samples. Here we report cryo-electron microscopy and negative-staining electron tomography approaches to image, and three-dimensionally reconstruct a single DNA-nanogold conjugate, an 84-bp double-stranded DNA with two 5-nm nanogold particles for potential substrates in plasmon-coupling experiments. By individual-particle electron tomography reconstruction, we obtain 14 density maps at ∼2-nm resolution. Using these maps as constraints, we derive 14 conformations of dsDNA by molecular dynamics simulations. The conformational variation is consistent with that from liquid solution, suggesting that individual-particle electron tomography could be an expected approach to study DNA-assembling and flexible protein structure and dynamics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2948.map.gz emd_2948.map.gz | 83.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2948-v30.xml emd-2948-v30.xml emd-2948.xml emd-2948.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2948.tif emd_2948.tif | 145.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2948 http://ftp.pdbj.org/pub/emdb/structures/EMD-2948 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2948 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2948 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2948.map.gz / 形式: CCP4 / 大きさ: 100.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2948.map.gz / 形式: CCP4 / 大きさ: 100.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of one particle of DNA-nanogold dimer by using individual-particle electron tomography | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.88 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Two 5-nm nanogolds bound to 84-base pair double-stranded DNA
| 全体 | 名称: Two 5-nm nanogolds bound to 84-base pair double-stranded DNA |
|---|---|
| 要素 |
|
-超分子 #1000: Two 5-nm nanogolds bound to 84-base pair double-stranded DNA
| 超分子 | 名称: Two 5-nm nanogolds bound to 84-base pair double-stranded DNA タイプ: sample / ID: 1000 詳細: 5 nm nanogold particles were stabilized via exchanging with bis-(p-sulfonatophenyl) phenylphosphine (BSPP). DNA sequences modified with a 5 thiol moiety were PAGE purified. DNA thiolated at ...詳細: 5 nm nanogold particles were stabilized via exchanging with bis-(p-sulfonatophenyl) phenylphosphine (BSPP). DNA sequences modified with a 5 thiol moiety were PAGE purified. DNA thiolated at the 5 end was re-suspended in buffer (10mM Tris pH 8, 0.5mM EDTA). Nanogold particles and DNA were combined at a stoichiometric ratio of 1:2 in the presence of a reducing agent. Monoconjugates formed were separated by anion exchange HPLC, and the fractions concentrated by an Amicon Ultra spin filter, MW 100,000 (EMD Millipore Corp, Billerica, MA). Twenty microliters of nanogold monoconjugates, each containing complementary strands of DNA, were combined stoichiometrically as determined by absorption at 520 nm and allowed to react overnight at room temperature. The dimers were purified from unreacted monoconjugates by agarose gel electrophoresis. 集合状態: Dimer / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 52 KDa / 理論値: 52 KDa / 手法: Calculated from its sequence |
-分子 #1: double-stranded DNA
| 分子 | 名称: double-stranded DNA / タイプ: dna / ID: 1 / Name.synonym: dsDNA 詳細: Two 5-nm nanogold bound to 84-base pair double-stranded DNA 分類: DNA / Structure: DOUBLE HELIX / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 実験値: 52 KDa / 理論値: 52 KDa |
| 配列 | 文字列: CCGGCGGCCC AGGTGTATCA GTGTTCGTTG CAAGCTCCAA CATCTGAGTA CCACGCATAC TATACTTGAA ATATCCGCGC CCGG |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 1X Dulbeccos phosphate-buffered saline (Invitrogen, La Jolla, CA), 2.7 mM KCl, 1.46 mM KH2PO4, 136.9 mM NaCl, and 8.1 mM Na2HPO4 |
| 染色 | タイプ: NEGATIVE 詳細: EM Specimens were prepared by optimized negative-staining EM specimen preparation protocol as described (Zhang L. and Ren G, Journal of Lipid Research, (2010) 51, 1228-1236 and (2011) 52, 175- ...詳細: EM Specimens were prepared by optimized negative-staining EM specimen preparation protocol as described (Zhang L. and Ren G, Journal of Lipid Research, (2010) 51, 1228-1236 and (2011) 52, 175-84). In brief, nanogold-DNA dimer was diluted to 0.02 mg/ml with DPBS. Aliquots (about 4ul) were applied to the 200 mesh glow-discharged thin carbon-coated EM grids (Cu-200CN, Pacific Grid-Tech, USA). The grid was washed by deionized water for three times, and then washed by 1% uranyl formate for three times before blotting to drying. |
| グリッド | 詳細: 200 mesh glow-discharged thin carbon-coated EM grids (Cu-200CN, Pacific Grid-Tech, USA) |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | ZEISS LIBRA120PLUS |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 0.5 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 125000 Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 0.5 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 125000 |
| 特殊光学系 | エネルギーフィルター - 名称: ZEISS エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダー: Gatan / 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° / Tilt series - Axis1 - Angle increment: 1.5 ° |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 125,000 times magnification |
| 詳細 | tilt step is 1.5 degree |
| 日付 | 2012年8月15日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 81 / 平均電子線量: 1000 e/Å2 / ビット/ピクセル: 16 |
- 画像解析
画像解析
| CTF補正 | 詳細: TOMOCTF |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 14.7 Å / 解像度の算出法: OTHER ソフトウェア - 名称: IPET, FETR, Spider, IMOD, EMAN, and, EMAN2 詳細: Map was reconstructed by individual-particle electron tomography (IPET)and Focus ET Reconstruction Algorithm 使用した粒子像数: 81 |
| 詳細 | Micrographs were initially aligned together with the IMOD software package. The CTF was then corrected by TOMOCTF. The tilt series of particles in square windows of 512 pixels (~48 nm) were semi-automatically tracked and windowed by individual-particle electron tomography (IPET) software{eulerAnglesDetails}: Tomography tilt angle from -60 to 60 in step of 1.5 |
 ムービー
ムービー コントローラー
コントローラー