+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2940 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural characterization of the Olfactomedin-1 disulfide-linked tetramer | |||||||||
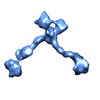
 マップデータ マップデータ | Filtered subtomogram of Olfactomedin-1 tetramer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Olfactomedin-1 (Olfm1) /  neurobiology (神経科学) / neurobiology (神経科学) /  tetramer (四量体) / tetramer (四量体) /  X-ray crystallography (X線回折) / X-ray crystallography (X線回折) /  small-angle X-ray scattering (SAXS) (X線小角散乱) / small-angle X-ray scattering (SAXS) (X線小角散乱) /  electron tomography / analytical ultracentrifugation (AUC) / electron tomography / analytical ultracentrifugation (AUC) /  coiled coil (コイルドコイル) / coiled coil (コイルドコイル) /  calcium (カルシウム) / calcium (カルシウム) /  disulfide (ジスルフィド) disulfide (ジスルフィド) | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  電子線トモグラフィー法 / 電子線トモグラフィー法 /  ネガティブ染色法 ネガティブ染色法 | |||||||||
 データ登録者 データ登録者 | Sharp TH / Pronker MF / Bos TG / Thies-Weesie DM / Janssen BJC | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2015 ジャーナル: J Biol Chem / 年: 2015タイトル: Olfactomedin-1 Has a V-shaped Disulfide-linked Tetrameric Structure. 著者: Matti F Pronker / Trusanne G A A Bos / Thomas H Sharp / Dominique M E Thies-Weesie / Bert J C Janssen /  要旨: Olfactomedin-1 (Olfm1; also known as noelin and pancortin) is a member of the olfactomedin domain-containing superfamily and a highly expressed neuronal glycoprotein important for nervous system ...Olfactomedin-1 (Olfm1; also known as noelin and pancortin) is a member of the olfactomedin domain-containing superfamily and a highly expressed neuronal glycoprotein important for nervous system development. It binds a number of secreted proteins and cell surface-bound receptors to induce cell signaling processes. Using a combined approach of x-ray crystallography, solution scattering, analytical ultracentrifugation, and electron microscopy we determined that full-length Olfm1 forms disulfide-linked tetramers with a distinctive V-shaped architecture. The base of the "V" is formed by two disulfide-linked dimeric N-terminal domains. Each of the two V legs consists of a parallel dimeric disulfide-linked coiled coil with a C-terminal β-propeller dimer at the tips. This agrees with our crystal structure of a C-terminal coiled-coil segment and β-propeller combination (Olfm1(coil-Olf)) that reveals a disulfide-linked dimeric arrangement with the β-propeller top faces in an outward exposed orientation. Similar to its family member myocilin, Olfm1 is stabilized by calcium. The dimer-of-dimers architecture suggests a role for Olfm1 in clustering receptors to regulate signaling and sheds light on the conformation of several other olfactomedin domain family members. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2940.map.gz emd_2940.map.gz | 377.1 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2940-v30.xml emd-2940-v30.xml emd-2940.xml emd-2940.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  2940.png 2940.png | 67.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2940 http://ftp.pdbj.org/pub/emdb/structures/EMD-2940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2940 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2940.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2940.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Filtered subtomogram of Olfactomedin-1 tetramer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.57 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Olfactomedin-1 disulfide-linked tetramer
| 全体 | 名称: Olfactomedin-1 disulfide-linked tetramer |
|---|---|
| 要素 |
|
-超分子 #1000: Olfactomedin-1 disulfide-linked tetramer
| 超分子 | 名称: Olfactomedin-1 disulfide-linked tetramer / タイプ: sample / ID: 1000 / 集合状態: Tetramer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 242 KDa / 理論値: 256 KDa / 手法: AUC |
-超分子 #1: Olfactomedin-1
| 超分子 | 名称: Olfactomedin-1 / タイプ: organelle_or_cellular_component / ID: 1 / Name.synonym: Olfm1, noelin, pancortin 詳細: Purified recombinant full-length glycoslated Olfactomedin-1 tetramers were negatively stained with uranyl formate. コピー数: 1 / 集合状態: Tetramer / 組換発現: Yes |
|---|---|
| Ref INTERPRO | divclassse qspanoncli ckpopupspa nclassgree n(this)spandata popltspanc lassquotlo adingbarqu otgtltimgs rcquotimgl oadinggifq uotdecodin gquotasync quotgtltsp angtdataur lajaxphp?m odetaxoamp ... divclassse qspanoncli ckpopupspa nclassgree n(this)spandata popltspanc lassquotlo adingbarqu otgtltimgs rcquotimgl oadinggifq uotdecodin gquotasync quotgtltsp angtdataur lajaxphp?m odetaxoamp kIPR022082 ampajax1cl asspoptrgi IPR022082i spandiv |
| Ref INTERPRO | divclassse qspanoncli ckpopupspa nclassgree n(this)spandata popltspanc lassquotlo adingbarqu otgtltimgs rcquotimgl oadinggifq uotdecodin gquotasync quotgtltsp angtdataur lajaxphp?m odetaxoamp ... divclassse qspanoncli ckpopupspa nclassgree n(this)spandata popltspanc lassquotlo adingbarqu otgtltimgs rcquotimgl oadinggifq uotdecodin gquotasync quotgtltsp angtdataur lajaxphp?m odetaxoamp kIPR003112 ampajax1cl asspoptrgi IPR003112i spandiv |
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) / 別称: House Mouse / 組織: brain / 細胞: neuron / Organelle: Secretory pathway / 細胞中の位置: Secreted (ER/golgi/extracellular) Mus musculus (ハツカネズミ) / 別称: House Mouse / 組織: brain / 細胞: neuron / Organelle: Secretory pathway / 細胞中の位置: Secreted (ER/golgi/extracellular) |
| 分子量 | 実験値: 242 KDa / 理論値: 256 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293ES / 組換プラスミド: pUPE107.03 Homo sapiens (ヒト) / 組換細胞: HEK293ES / 組換プラスミド: pUPE107.03 |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.065 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 150 mM NaCl, 20 mM HEPES |
| 染色 | タイプ: NEGATIVE 詳細: Full-length Olfm1 was adsorbed to grids for 30 sec. Grids were briefly washed with water and then stained for 30 sec with a freshly prepared filtered 2% uranyl formate solution. |
| グリッド | 詳細: Carbon-coated mesh copper grids glow discharged for 15 sec |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 30000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt series - Axis1 - Min angle: -58 ° / Tilt series - Axis1 - Max angle: 58 ° / Tilt series - Axis1 - Angle increment: 2 ° |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 30,000 times magnification |
| 詳細 | Weak beam illumination |
| 日付 | 2015年2月12日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 58 / ビット/ピクセル: 32 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: IMOD, each tilt image |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / ソフトウェア - 名称: IMOD, EMAN2 詳細: Sub-tomogram particles were manually picked using e2spt_boxer.py from EMAN2. Each particle was normalized and masked with a sharp spherical mask to remove background density not associated ...詳細: Sub-tomogram particles were manually picked using e2spt_boxer.py from EMAN2. Each particle was normalized and masked with a sharp spherical mask to remove background density not associated with the protein. Particles were then filtered to 20 A with a low-pass Gaussian filter, before a tight mask was applied to the remaining density using e2proc3d.py from EMAN2. 使用した粒子像数: 57 |
| 詳細 | Tilt series were collected from -58 to 58 degrees in 2 degree increments. The first and last image was discarded. CTF correction was performed on each projection |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Each coiled-coil dimer of the tetramer was fitted as a rigid body. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー