+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1399 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions. | |||||||||
 マップデータ マップデータ | 3d MAP OF SUB-SET OF HEPATITIS B VIRUS Particles (COMPACT PARTICLES; grouped in the same class) | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |    Hepatitis B virus (B 型肝炎ウイルス) Hepatitis B virus (B 型肝炎ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Seitz S / Urban S / Antoni C / Bottcher B | |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2007 ジャーナル: EMBO J / 年: 2007タイトル: Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions. 著者: Stefan Seitz / Stephan Urban / Christoph Antoni / Bettina Böttcher /  要旨: Hepatitis B virus (HBV) is a major human pathogen causing about 750,000 deaths per year. The virion consists of a nucleocapsid and an envelope formed by lipids, and three integral membrane proteins. ...Hepatitis B virus (HBV) is a major human pathogen causing about 750,000 deaths per year. The virion consists of a nucleocapsid and an envelope formed by lipids, and three integral membrane proteins. Although we have detailed structural insights into the organization of the HBV core, the arrangement of the envelope in virions and its interaction with the nucleocapsid is elusive. Here we show the ultrastructure of hepatitis B virions purified from patient serum. We identified two morphological phenotypes, which appear as compact and gapped particles with nucleocapsids in distinguishable conformations. The overall structures of these nucleocapsids resemble recombinant cores with two alpha-helical spikes per asymmetric unit. At the charged tips the spikes are contacted by defined protrusions of the envelope proteins, probably via electrostatic interactions. The HBV envelope in the two morphotypes is to some extent variable, but the surface proteins follow a general packing scheme with up to three surface protein dimers per asymmetric unit. The variability in the structure of the envelope indicates that the nucleocapsid does not firmly constrain the arrangement of the surface proteins, but provides a general template for the packing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1399.map.gz emd_1399.map.gz | 527.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1399-v30.xml emd-1399-v30.xml emd-1399.xml emd-1399.xml | 8.4 KB 8.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1399.gif 1399.gif | 11 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1399 http://ftp.pdbj.org/pub/emdb/structures/EMD-1399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1399 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1399.map.gz / 形式: CCP4 / 大きさ: 955.1 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1399.map.gz / 形式: CCP4 / 大きさ: 955.1 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3d MAP OF SUB-SET OF HEPATITIS B VIRUS Particles (COMPACT PARTICLES; grouped in the same class) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 8.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
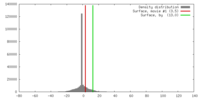
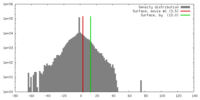
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Hepatitis B virus
| 全体 | 名称:    Hepatitis B virus (B 型肝炎ウイルス) Hepatitis B virus (B 型肝炎ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1000: Hepatitis B virus
| 超分子 | 名称: Hepatitis B virus / タイプ: sample / ID: 1000 / 詳細: virus purified from patients serum / Number unique components: 1 |
|---|
-超分子 #1: Hepatitis B virus
| 超分子 | 名称: Hepatitis B virus / タイプ: virus / ID: 1 / Name.synonym: HBV / NCBI-ID: 10407 / 生物種: Hepatitis B virus / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No / Syn species name: HBV |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | 詳細: 400 mesh copper rhodium grids (Maxtaform), |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 93 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: self made 手法: automatic blot for 1-2 seconds from both sides before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM120T |
|---|---|
| 電子線 | 加速電圧: 100 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 50000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 6.3 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 52000 Bright-field microscopy / Cs: 6.3 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダー: Side entry liquid nitrogen-cooled cryo specimen holder 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 178 K |
| アライメント法 | Legacy - 非点収差: corrected at 200,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 21 µm / 実像数: 74 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 8 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each Particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / アルゴリズム: OTHER / ソフトウェア - 名称: MRC / 使用した粒子像数: 15 ) / アルゴリズム: OTHER / ソフトウェア - 名称: MRC / 使用した粒子像数: 15 |
| 詳細 | particles were selected manually |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)