+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1376 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Averaging tens to hundreds of icosahedral particle images to resolve protein secondary structure elements using a Multi-Path Simulated Annealing optimization algorithm. | |||||||||
 マップデータ マップデータ | This is a P3A subunit of a RDV (Rice Dwarf Virus) map. It was reconstructed from the best 284 particles. For the full map, see accession code: EMD-1375. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |    Rice dwarf virus (イネ萎縮ウイルス) Rice dwarf virus (イネ萎縮ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.9 Å クライオ電子顕微鏡法 / 解像度: 7.9 Å | |||||||||
 データ登録者 データ登録者 | Liu X / Jiang W / Jakana J / Chiu W | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2007 ジャーナル: J Struct Biol / 年: 2007タイトル: Averaging tens to hundreds of icosahedral particle images to resolve protein secondary structure elements using a Multi-Path Simulated Annealing optimization algorithm. 著者: Xiangan Liu / Wen Jiang / Joanita Jakana / Wah Chiu /  要旨: Accurately determining a cryoEM particle's alignment parameters is crucial to high resolution single particle 3-D reconstruction. We developed Multi-Path Simulated Annealing, a Monte-Carlo type of ...Accurately determining a cryoEM particle's alignment parameters is crucial to high resolution single particle 3-D reconstruction. We developed Multi-Path Simulated Annealing, a Monte-Carlo type of optimization algorithm, for globally aligning the center and orientation of a particle simultaneously. A consistency criterion was developed to ensure the alignment parameters are correct and to remove some bad particles from a large pool of images of icosahedral particles. Without using any a priori model, this procedure is able to reconstruct a structure from a random initial model. Combining the procedure above with a new empirical double threshold particle selection method, we are able to pick tens of best quality particles to reconstruct a subnanometer resolution map from scratch. Using the best 62 particles of rice dwarf virus, the reconstruction reached 9.6A resolution at which four helices of the P3A subunit of RDV are resolved. Furthermore, with the 284 best particles, the reconstruction is improved to 7.9A resolution, and 21 of 22 helices and six of seven beta sheets are resolved. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1376.map.gz emd_1376.map.gz | 505.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1376-v30.xml emd-1376-v30.xml emd-1376.xml emd-1376.xml | 8.8 KB 8.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1376.gif 1376.gif | 50.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1376 http://ftp.pdbj.org/pub/emdb/structures/EMD-1376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1376 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1375C  1377C  1378C  1379C  1380C  1381C  1382C  1383C  1384C  1385C  1386C  1387C  1388C  1389C  1390C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1376.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1376.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a P3A subunit of a RDV (Rice Dwarf Virus) map. It was reconstructed from the best 284 particles. For the full map, see accession code: EMD-1375. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.48906 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
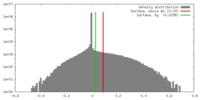
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rice Dwarf Virus
| 全体 | 名称:    Rice Dwarf Virus (イネ萎縮ウイルス) Rice Dwarf Virus (イネ萎縮ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1000: Rice Dwarf Virus
| 超分子 | 名称: Rice Dwarf Virus / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 50 MDa |
-超分子 #1: Rice dwarf virus
| 超分子 | 名称: Rice dwarf virus / タイプ: virus / ID: 1 / NCBI-ID: 10991 / 生物種: Rice dwarf virus / ウイルスタイプ: OTHER / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 別称: PLANTAE(HIGHER PLANTS) |
| ウイルス殻 | Shell ID: 1 / 名称: Inner Shell / 直径: 550 Å / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 27 % / チャンバー内温度: 111 K |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 4000 |
|---|---|
| 電子線 | 加速電圧: 400 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 49495 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 4.1 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 4.1 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Gatan / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 109 K |
| 詳細 | Microscope:JEOL 4000 |
| 日付 | 1999年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 4 µm / 実像数: 100 / 平均電子線量: 13 e/Å2 / ビット/ピクセル: 32 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.9 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Multi-path SA ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.9 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Multi-path SA詳細: The 5 alignment parameters (2 for center and 3 for orientation) were searched simultaneously for each particle image. The search is executed by a new optimizaiton algorithm, multi-path ...詳細: The 5 alignment parameters (2 for center and 3 for orientation) were searched simultaneously for each particle image. The search is executed by a new optimizaiton algorithm, multi-path simulated annealing, based on Cross Common Lines. There were 4200 good particles discriminated from 4865 raw particles by a Consistency Criterion. The best 284 particles were empirically selected by a double threshold method from the 4200 good particles. The map's resolution was measured against the Known X-ray structure (PDB ID:1UF2). 使用した粒子像数: 284 |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)