+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10129 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of TMV in water | |||||||||
 マップデータ マップデータ | B-factor sharpened and locally filtered | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Tobacco mosaic virus-like, coat protein / Tobacco mosaic virus-like, coat protein superfamily / Virus coat protein (TMV like) / helical viral capsid / structural molecule activity / identical protein binding /  カプシド カプシド 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |    TMV (タバコモザイクウイルス) / TMV (タバコモザイクウイルス) /   Tobacco mosaic virus (strain vulgare) (タバコモザイクウイルス) Tobacco mosaic virus (strain vulgare) (タバコモザイクウイルス) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 1.9 Å クライオ電子顕微鏡法 / 解像度: 1.9 Å | |||||||||
 データ登録者 データ登録者 | Weis F / Beckers M / Sachse C | |||||||||
 引用 引用 |  ジャーナル: EMBO Rep / 年: 2019 ジャーナル: EMBO Rep / 年: 2019タイトル: Elucidation of the viral disassembly switch of tobacco mosaic virus. 著者: Felix Weis / Maximilian Beckers / Iris von der Hocht / Carsten Sachse /  要旨: Stable capsid structures of viruses protect viral RNA while they also require controlled disassembly for releasing the viral genome in the host cell. A detailed understanding of viral disassembly ...Stable capsid structures of viruses protect viral RNA while they also require controlled disassembly for releasing the viral genome in the host cell. A detailed understanding of viral disassembly processes and the involved structural switches is still lacking. This process has been extensively studied using tobacco mosaic virus (TMV), and carboxylate interactions are assumed to play a critical part in this process. Here, we present two cryo-EM structures of the helical TMV assembly at 2.0 and 1.9 Å resolution in conditions of high Ca concentration at low pH and in water. Based on our atomic models, we identify the conformational details of the disassembly switch mechanism: In high Ca /acidic pH environment, the virion is stabilized between neighboring subunits through carboxyl groups E95 and E97 in close proximity to a Ca binding site that is shared between two subunits. Upon increase in pH and lower Ca levels, mutual repulsion of the E95/E97 pair and Ca removal destabilize the network of interactions between adjacent subunits at lower radius and release the switch for viral disassembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10129.map.gz emd_10129.map.gz | 395 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10129-v30.xml emd-10129-v30.xml emd-10129.xml emd-10129.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10129.png emd_10129.png | 322.7 KB | ||
| マスクデータ |  emd_10129_msk_1.map emd_10129_msk_1.map | 437.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10129_additional.map.gz emd_10129_additional.map.gz emd_10129_half_map_1.map.gz emd_10129_half_map_1.map.gz emd_10129_half_map_2.map.gz emd_10129_half_map_2.map.gz | 16.4 MB 351 MB 351 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10129 http://ftp.pdbj.org/pub/emdb/structures/EMD-10129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10129 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6saeMC  6sagC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10305 (タイトル: Cryo-EM structure of TMV in water / Data size: 6.8 EMPIAR-10305 (タイトル: Cryo-EM structure of TMV in water / Data size: 6.8 Data #1: Raw movies of the TMV in water sample, with corresponding start-end coordinates files and gain reference file [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10129.map.gz / 形式: CCP4 / 大きさ: 437.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10129.map.gz / 形式: CCP4 / 大きさ: 437.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | B-factor sharpened and locally filtered | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.638 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
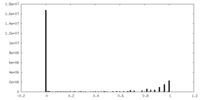
| ファイル |  emd_10129_msk_1.map emd_10129_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Confidence map
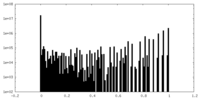
| ファイル | emd_10129_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Confidence map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 1
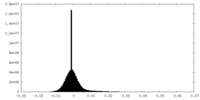
| ファイル | emd_10129_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 2
| ファイル | emd_10129_half_map_2.map | ||||||||||||
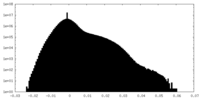
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tobacco mosaic virus (strain vulgare)
| 全体 | 名称:   Tobacco mosaic virus (strain vulgare) (タバコモザイクウイルス) Tobacco mosaic virus (strain vulgare) (タバコモザイクウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Tobacco mosaic virus (strain vulgare)
| 超分子 | 名称: Tobacco mosaic virus (strain vulgare) / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 / NCBI-ID: 12243 / 生物種: Tobacco mosaic virus (strain vulgare) / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Nicotiana tabacum (タバコ) Nicotiana tabacum (タバコ) |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 詳細: Residues 154-158 are flexible and were not modelled. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    TMV (タバコモザイクウイルス) / 株: vulgare TMV (タバコモザイクウイルス) / 株: vulgare |
| 分子量 | 理論値: 17.531463 KDa |
| 配列 | 文字列: (ACE)SYSITTPSQ FVFLSSAWAD PIELINLCTN ALGNQFQTQQ ARTVVQRQFS EVWKPSPQVT VRFPDSDFKV YRYNAV LDP LVTALLGAFD TRNRIIEVEN QANPTTAETL DATRRVDDAT VAIRSAINNL IVELIRGTGS YNRSSFESSS GLVWTSG PA T |
-分子 #2: RNA (5'-R(P*GP*AP*A)-3')
| 分子 | 名称: RNA (5'-R(P*GP*AP*A)-3') / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:    TMV (タバコモザイクウイルス) TMV (タバコモザイクウイルス) |
| 分子量 | 理論値: 958.66 Da |
| 配列 | 文字列: GAA |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 92 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 22 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 詳細: pure water |
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 詳細: Pelco Easyglow, factory settings |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.35 µm / 最小 デフォーカス(公称値): 0.15 µm / 倍率(公称値): 215000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.35 µm / 最小 デフォーカス(公称値): 0.15 µm / 倍率(公称値): 215000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-20 / 撮影したグリッド数: 1 / 実像数: 62 / 平均露光時間: 5.0 sec. / 平均電子線量: 30.8 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| Segment selection | 選択した数: 21709 / ソフトウェア - 名称: SPRING |
|---|---|
| CTF補正 | ソフトウェア - 名称: Gctf |
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: RELION (ver. 3) |
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 1.406 Å 想定した対称性 - らせんパラメータ - ΔΦ: 22.038 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 1.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3) / 使用した粒子像数: 21598 |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X