+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8dit | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
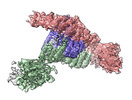
| タイトル | Cryo-EM structure of a HOPS core complex containing Vps33, Vps16, and Vps18 | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード |  PROTEIN TRANSPORT / PROTEIN TRANSPORT /  HOPS / membrane tethering / SNAREs / HOPS / membrane tethering / SNAREs /  membrane fusion / endolysosomal trafficking membrane fusion / endolysosomal trafficking | ||||||||||||
| 生物種 |   Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) | ||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.1 Å クライオ電子顕微鏡法 / 解像度: 5.1 Å | ||||||||||||
 データ登録者 データ登録者 | Port, S.A. / Farrell, P.D. / Jeffrey, P.D. / DiMaio, F. / Hughson, F.M. | ||||||||||||
| 資金援助 |  ドイツ, ドイツ,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of the HOPS core complex and its implication for SNARE assembly 著者: Port, S.A. / Farrell, P.D. / Jeffrey, P.D. / DiMaio, F. / Hughson, F.M. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8dit.cif.gz 8dit.cif.gz | 429.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8dit.ent.gz pdb8dit.ent.gz | 342 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8dit.json.gz 8dit.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/di/8dit https://data.pdbj.org/pub/pdb/validation_reports/di/8dit ftp://data.pdbj.org/pub/pdb/validation_reports/di/8dit ftp://data.pdbj.org/pub/pdb/validation_reports/di/8dit | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  液胞 液胞分子量: 77119.617 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Chaetomium thermophilum (菌類) / プラスミド: pQLinkH-Vps33:Vps16 / 詳細 (発現宿主): co-expression plasmid / 発現宿主: Chaetomium thermophilum (菌類) / プラスミド: pQLinkH-Vps33:Vps16 / 詳細 (発現宿主): co-expression plasmid / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21 codon+ Escherichia coli (大腸菌) / 株 (発現宿主): BL21 codon+ |
|---|---|
| #2: タンパク質 |  液胞 液胞分子量: 94204.828 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Chaetomium thermophilum (菌類) / プラスミド: pQLinkH-Vps33:Vps16 / 詳細 (発現宿主): co-expression plasmid / 発現宿主: Chaetomium thermophilum (菌類) / プラスミド: pQLinkH-Vps33:Vps16 / 詳細 (発現宿主): co-expression plasmid / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21 codon+ Escherichia coli (大腸菌) / 株 (発現宿主): BL21 codon+ |
| #3: タンパク質 |  液胞 液胞分子量: 108527.570 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Chaetomium thermophilum (菌類) / プラスミド: pQLinkN-Vps18 / 発現宿主: Chaetomium thermophilum (菌類) / プラスミド: pQLinkN-Vps18 / 発現宿主:   Escherichia coli (大腸菌) / Variant (発現宿主): C43 Escherichia coli (大腸菌) / Variant (発現宿主): C43 |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 |
| ||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||
| 緩衝液 | pH: 7.4 | ||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||
| 試料 | 濃度: 0.08 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||||||
| 試料支持 | 詳細: PELCO Easiglow, 10 mA/ 10 s discharge/ 30 s hold / グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: UltrAuFoil R1.2/1.3 | ||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 298 K 詳細: Blotting parameters: 30 seconds waiting time, blotting force 1, blot total 1 |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 2000 nm / Cs Bright-field microscopy / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 2000 nm / Cs : 0.03 mm : 0.03 mm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 5.6 sec. / 電子線照射量: 50 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 3590 |
| 電子光学装置 | エネルギーフィルター名称 : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
| 画像スキャン | 横: 3838 / 縦: 3710 / 動画フレーム数/画像: 32 / 利用したフレーム数/画像: 1-32 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 4092244 / 詳細: blob picker 60-280A (circular) | ||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 5.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 154917 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 精密化 | 最高解像度: 5.1 Å |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj