+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8bgw | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of quinol-dependent Nitric Oxide Reductase (qNOR) from Alcaligenes xylosoxidans at 2.2 A resolution | |||||||||
 要素 要素 | Nitric oxide reductase subunit B | |||||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / quinol-dependent Nitric Oxide Reductase / proton transfer / MEMBRANE PROTEIN (膜タンパク質) / quinol-dependent Nitric Oxide Reductase / proton transfer /  quinol binding (ヒドロキノン) / quinol binding (ヒドロキノン) /  ubiquinol oxidase ubiquinol oxidase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 nitric oxide reductase (cytochrome c) / nitric oxide reductase activity / nitric oxide reductase (cytochrome c) / nitric oxide reductase activity /  cytochrome-c oxidase activity / cytochrome-c oxidase activity /  細胞呼吸 / 細胞呼吸 /  heme binding / heme binding /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Achromobacter xylosoxidans (バクテリア) Achromobacter xylosoxidans (バクテリア) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.2 Å クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Flynn, A. / Antonyuk, S.V. / Eady, R.R. / Muench, S.P. / Hasnain, S.S. | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: A 2.2 Å cryoEM structure of a quinol-dependent NO Reductase shows close similarity to respiratory oxidases. 著者: Alex J Flynn / Svetlana V Antonyuk / Robert R Eady / Stephen P Muench / S Samar Hasnain /  要旨: Quinol-dependent nitric oxide reductases (qNORs) are considered members of the respiratory heme-copper oxidase superfamily, are unique to bacteria, and are commonly found in pathogenic bacteria where ...Quinol-dependent nitric oxide reductases (qNORs) are considered members of the respiratory heme-copper oxidase superfamily, are unique to bacteria, and are commonly found in pathogenic bacteria where they play a role in combating the host immune response. qNORs are also essential enzymes in the denitrification pathway, catalysing the reduction of nitric oxide to nitrous oxide. Here, we determine a 2.2 Å cryoEM structure of qNOR from Alcaligenes xylosoxidans, an opportunistic pathogen and a denitrifying bacterium of importance in the nitrogen cycle. This high-resolution structure provides insight into electron, substrate, and proton pathways, and provides evidence that the quinol binding site not only contains the conserved His and Asp residues but also possesses a critical Arg (Arg720) observed in cytochrome bo, a respiratory quinol oxidase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8bgw.cif.gz 8bgw.cif.gz | 332 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8bgw.ent.gz pdb8bgw.ent.gz | 265.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8bgw.json.gz 8bgw.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bg/8bgw https://data.pdbj.org/pub/pdb/validation_reports/bg/8bgw ftp://data.pdbj.org/pub/pdb/validation_reports/bg/8bgw ftp://data.pdbj.org/pub/pdb/validation_reports/bg/8bgw | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  16041MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSアンサンブル: (詳細: Local NCS retraints between domains: 1 2) |
- 要素
要素
-タンパク質 / 糖 , 2種, 4分子 AB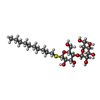

| #1: タンパク質 | 分子量: 84724.867 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Achromobacter xylosoxidans (バクテリア) Achromobacter xylosoxidans (バクテリア)遺伝子: norB_1, ERS451415_02175, I6G72_25885 / プラスミド: PET-26B (+) / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): C41 Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): C41参照: UniProt: A0A0D6H8R3,  nitric oxide reductase (cytochrome c) nitric oxide reductase (cytochrome c)#5: 糖 | |
|---|
-非ポリマー , 6種, 472分子 


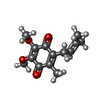







| #2: 化合物 | ChemComp-HEM /  Heme B Heme B#3: 化合物 |  鉄 鉄#4: 化合物 | #6: 化合物 | #7: 化合物 | ChemComp-LOP / ( #8: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: quinol-dependent Nitric Oxide Reductase / タイプ: CELL / 詳細: quinol-dependent Nitric Oxide Reductase / Entity ID: #1 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.84651 MDa / 実験値: NO |
| 由来(天然) | 生物種:   Achromobacter xylosoxidans (バクテリア) Achromobacter xylosoxidans (バクテリア) |
| 由来(組換発現) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 緩衝液 | pH: 7 / 詳細: 50mM Tris-HCl, 150mM NaCl, 0.05% DTM |
| 緩衝液成分 | 濃度: 50 mM 名称: Tris-HCL  トリスヒドロキシメチルアミノメタン トリスヒドロキシメチルアミノメタン式: Tris-HCL  トリスヒドロキシメチルアミノメタン トリスヒドロキシメチルアミノメタン |
| 試料 | 濃度: 3 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES / 詳細: Sample was monodisperse : YES / 詳細: Sample was monodisperse |
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K 詳細: Sample at 3 mg/mL was applied to glow-discharged Quantifoil Au R1.2/1.3 holey carbo grids |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 130000 X / 最大 デフォーカス(公称値): 2700 nm / 最小 デフォーカス(公称値): 900 nm / Cs Bright-field microscopy / 倍率(公称値): 130000 X / 最大 デフォーカス(公称値): 2700 nm / 最小 デフォーカス(公称値): 900 nm / Cs : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER 最高温度: 114 K / 最低温度: 80 K |
| 撮影 | 平均露光時間: 6.11 sec. / 電子線照射量: 34.9 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 5466 |
| 電子光学装置 | エネルギーフィルター名称 : TFS Selectris / エネルギーフィルタースリット幅: 10 eV : TFS Selectris / エネルギーフィルタースリット幅: 10 eV |
| 画像スキャン | 横: 4096 / 縦: 4096 |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0267 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 3000000 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C2 (2回回転対称 : C2 (2回回転対称 ) ) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 2.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 404950 / アルゴリズム: FOURIER SPACE / クラス平均像の数: 2 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 47 / プロトコル: RIGID BODY FIT / 空間: RECIPROCAL / Target criteria: Cross-correlation coefficient | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 最高解像度: 2.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj







