| 登録構造単位 | A: Insulin-like growth factor 2 mRNA-binding protein 3
B: RNA (5'-R(P*CP*AP*CP*AP*CP*AP*C)-3')
C: Insulin-like growth factor 2 mRNA-binding protein 3
D: RNA (5'-R(P*CP*AP*CP*AP*CP*AP*C)-3')
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 40,392 | 4 |
|---|
| ポリマ- | 40,392 | 4 |
|---|
| 非ポリマー | 0 | 0 |
|---|
| 水 | 775 | 43 |
|---|
|
|---|
| 1 | A: Insulin-like growth factor 2 mRNA-binding protein 3
B: RNA (5'-R(P*CP*AP*CP*AP*CP*AP*C)-3')
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 20,196 | 2 |
|---|
| ポリマ- | 20,196 | 2 |
|---|
| 非ポリマー | 0 | 0 |
|---|
| 水 | 36 | 2 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
|
|---|
| 2 | C: Insulin-like growth factor 2 mRNA-binding protein 3
D: RNA (5'-R(P*CP*AP*CP*AP*CP*AP*C)-3')
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 20,196 | 2 |
|---|
| ポリマ- | 20,196 | 2 |
|---|
| 非ポリマー | 0 | 0 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
|
|---|
| 単位格子 | | Length a, b, c (Å) | 45.890, 73.716, 57.405 |
|---|
| Angle α, β, γ (deg.) | 90.000, 101.796, 90.000 |
|---|
| Int Tables number | 4 |
|---|
| Space group name H-M | P1211 |
|---|
| Space group name Hall | P2yb |
|---|
| Symmetry operation | #1: x,y,z
#2: -x,y+1/2,-z |
|---|
|
|---|
| 非結晶学的対称性 (NCS) | NCSドメイン: | ID | Ens-ID | 詳細 |
|---|
d_1ens_1(chain "A" and (resid 0 through 19 or resid 21 through 159))d_2ens_1(chain "C" and (resid 0 through 19 or resid 21 through 159))d_1ens_2chain "B"d_2ens_2| chain "D" | | | | | | | | | | | |
NCSドメイン領域: | Dom-ID | Component-ID | Ens-ID | Beg auth comp-ID | Beg label comp-ID | End auth comp-ID | End label comp-ID | Auth asym-ID | Label asym-ID | Auth seq-ID | Label seq-ID |
|---|
d_1| 1 | ens_1SERSERGLUGLUAA| 0 - 19 | 1 - 20 | d_1| 2 | ens_1VALVALALAALAAA| 21 - 159 | 22 - 160 | d_2| 1 | ens_1SERSERGLUGLUCC| 0 - 19 | 1 - 20 | d_2| 2 | ens_1VALVALALAALACC| 21 - 159 | 22 - 160 | d_1| 1 | ens_2AACCBB| 1 - 4 | 2 - 5 | d_2| 1 | ens_2AACCDD| 1 - 4 | 2 - 5 | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | | |
NCSアンサンブル:
NCS oper: | ID | Code | Matrix | ベクター |
|---|
| 1 | given(-0.450878705728, 0.00147247366442, 0.892584015397), (0.00163759382556, -0.999995591667, 0.00247687984515), (0.89258372773, 0.00257846245123, 0.45087430679)-16.7631968756, 24.7734969464, -28.1065211888| 2 | given(-0.38766228214, -0.0146519866947, 0.921685019023), (0.055465319135, -0.998432769409, 0.00745676494762), (0.920131269645, 0.0540122602391, 0.38786740307)| -16.4170433664, 24.7712301866, -28.6080295675 | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード IGF2BP3 / IGF2BP /
IGF2BP3 / IGF2BP /  IMP3 /
IMP3 /  RNA binding (リボ核酸) / m6A modification /
RNA binding (リボ核酸) / m6A modification /  RNA BINDING PROTEIN (RNA結合タンパク質) / RNA BINDING PROTEIN-RNA complex
RNA BINDING PROTEIN (RNA結合タンパク質) / RNA BINDING PROTEIN-RNA complex 機能・相同性情報
機能・相同性情報 P-body / mRNA 5'-UTR binding / cytoplasmic stress granule /
P-body / mRNA 5'-UTR binding / cytoplasmic stress granule /  regulation of translation /
regulation of translation /  nervous system development /
nervous system development /  遺伝子発現の調節 ...CRD-mediated mRNA stabilization / N6-methyladenosine-containing RNA reader activity / mRNA transport / mRNA 3'-UTR binding /
遺伝子発現の調節 ...CRD-mediated mRNA stabilization / N6-methyladenosine-containing RNA reader activity / mRNA transport / mRNA 3'-UTR binding /  P-body / mRNA 5'-UTR binding / cytoplasmic stress granule /
P-body / mRNA 5'-UTR binding / cytoplasmic stress granule /  regulation of translation /
regulation of translation /  nervous system development /
nervous system development /  遺伝子発現の調節 /
遺伝子発現の調節 /  細胞核 /
細胞核 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.5 Å
分子置換 / 解像度: 2.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7yew.cif.gz
7yew.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7yew.ent.gz
pdb7yew.ent.gz PDB形式
PDB形式 7yew.json.gz
7yew.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ye/7yew
https://data.pdbj.org/pub/pdb/validation_reports/ye/7yew ftp://data.pdbj.org/pub/pdb/validation_reports/ye/7yew
ftp://data.pdbj.org/pub/pdb/validation_reports/ye/7yew
 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体

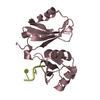
 要素
要素
 Mus musculus (ハツカネズミ) / 遺伝子: Igf2bp3, Vickz3 / 発現宿主:
Mus musculus (ハツカネズミ) / 遺伝子: Igf2bp3, Vickz3 / 発現宿主: 
 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q9CPN8
Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q9CPN8 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL18U1 / 波長: 0.97915 Å
/ ビームライン: BL18U1 / 波長: 0.97915 Å : 0.97915 Å / 相対比: 1
: 0.97915 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj

