+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7w1m | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
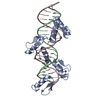
| タイトル | Cryo-EM structure of human cohesin-CTCF-DNA complex | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA /  Cohesin (コヒーシン) / NIPBL / Cohesin (コヒーシン) / NIPBL /  CTCF (CTCF) / CTCF (CTCF) /  DNA (デオキシリボ核酸) / chromosome folding / topologically associating domain / chromatin loops / DNA loop extrusion / sister chromatid cohesion / DNA (デオキシリボ核酸) / chromosome folding / topologically associating domain / chromatin loops / DNA loop extrusion / sister chromatid cohesion /  complex / complex /  ATPase (ATPアーゼ) / ATPase (ATPアーゼ) /  HEAT repeat protein (HEATリピート) / HEAT repeat protein (HEATリピート) /  DNA BINDING PROTEIN (DNA結合タンパク質) / DNA BINDING PROTEIN-DNA complex DNA BINDING PROTEIN (DNA結合タンパク質) / DNA BINDING PROTEIN-DNA complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報eye morphogenesis / external genitalia morphogenesis / gallbladder development / SMC loading complex / Scc2-Scc4 cohesin loading complex / ear morphogenesis / mitotic cohesin loading / response to DNA damage checkpoint signaling /  regulation of hair cycle / negative regulation of mitotic metaphase/anaphase transition ...eye morphogenesis / external genitalia morphogenesis / gallbladder development / SMC loading complex / Scc2-Scc4 cohesin loading complex / ear morphogenesis / mitotic cohesin loading / response to DNA damage checkpoint signaling / regulation of hair cycle / negative regulation of mitotic metaphase/anaphase transition ...eye morphogenesis / external genitalia morphogenesis / gallbladder development / SMC loading complex / Scc2-Scc4 cohesin loading complex / ear morphogenesis / mitotic cohesin loading / response to DNA damage checkpoint signaling /  regulation of hair cycle / negative regulation of mitotic metaphase/anaphase transition / chromatin insulator sequence binding / cohesin loader activity / positive regulation of sister chromatid cohesion / meiotic cohesin complex / Cohesin Loading onto Chromatin / maintenance of mitotic sister chromatid cohesion / regulation of hair cycle / negative regulation of mitotic metaphase/anaphase transition / chromatin insulator sequence binding / cohesin loader activity / positive regulation of sister chromatid cohesion / meiotic cohesin complex / Cohesin Loading onto Chromatin / maintenance of mitotic sister chromatid cohesion /  Establishment of Sister Chromatid Cohesion / forelimb morphogenesis / embryonic viscerocranium morphogenesis / establishment of meiotic sister chromatid cohesion / mitotic cohesin complex / Establishment of Sister Chromatid Cohesion / forelimb morphogenesis / embryonic viscerocranium morphogenesis / establishment of meiotic sister chromatid cohesion / mitotic cohesin complex /  cohesin complex / cohesin complex /  integrator complex / negative regulation of G2/M transition of mitotic cell cycle / uterus morphogenesis / regulation of centromeric sister chromatid cohesion / establishment of protein localization to chromatin / negative regulation of glial cell apoptotic process / regulation of developmental growth / integrator complex / negative regulation of G2/M transition of mitotic cell cycle / uterus morphogenesis / regulation of centromeric sister chromatid cohesion / establishment of protein localization to chromatin / negative regulation of glial cell apoptotic process / regulation of developmental growth /  ゲノム刷り込み / embryonic digestive tract morphogenesis / replication-born double-strand break repair via sister chromatid exchange / cellular response to X-ray / lateral element / positive regulation of neuron migration / ゲノム刷り込み / embryonic digestive tract morphogenesis / replication-born double-strand break repair via sister chromatid exchange / cellular response to X-ray / lateral element / positive regulation of neuron migration /  mediator complex binding / mediator complex binding /  chromo shadow domain binding / protein localization to chromosome, centromeric region / establishment of mitotic sister chromatid cohesion / chromatin looping / positive regulation of multicellular organism growth / metanephros development / positive regulation of ossification / digestive tract development / reciprocal meiotic recombination / embryonic forelimb morphogenesis / chromo shadow domain binding / protein localization to chromosome, centromeric region / establishment of mitotic sister chromatid cohesion / chromatin looping / positive regulation of multicellular organism growth / metanephros development / positive regulation of ossification / digestive tract development / reciprocal meiotic recombination / embryonic forelimb morphogenesis /  lncRNA binding / negative regulation of gene expression via chromosomal CpG island methylation / sister chromatid cohesion / face morphogenesis / negative regulation of interleukin-1 beta production / lncRNA binding / negative regulation of gene expression via chromosomal CpG island methylation / sister chromatid cohesion / face morphogenesis / negative regulation of interleukin-1 beta production /  microtubule motor activity / mitotic sister chromatid cohesion / stem cell population maintenance / dynein complex binding / microtubule motor activity / mitotic sister chromatid cohesion / stem cell population maintenance / dynein complex binding /  beta-tubulin binding / fat cell differentiation / mitotic spindle pole / outflow tract morphogenesis / beta-tubulin binding / fat cell differentiation / mitotic spindle pole / outflow tract morphogenesis /  regulation of DNA replication / mitotic sister chromatid segregation / somatic stem cell population maintenance / positive regulation of interleukin-10 production / regulation of DNA replication / mitotic sister chromatid segregation / somatic stem cell population maintenance / positive regulation of interleukin-10 production /  regulation of embryonic development / regulation of embryonic development /  chromosome, centromeric region / negative regulation of tumor necrosis factor production / chromosome, centromeric region / negative regulation of tumor necrosis factor production /  mitotic spindle assembly / developmental growth / localization / SUMOylation of DNA damage response and repair proteins / epigenetic regulation of gene expression / heart morphogenesis / condensed chromosome / protein localization to chromatin / Resolution of Sister Chromatid Cohesion / Meiotic synapsis / meiotic cell cycle / condensed nuclear chromosome / male germ cell nucleus / mitotic spindle assembly / developmental growth / localization / SUMOylation of DNA damage response and repair proteins / epigenetic regulation of gene expression / heart morphogenesis / condensed chromosome / protein localization to chromatin / Resolution of Sister Chromatid Cohesion / Meiotic synapsis / meiotic cell cycle / condensed nuclear chromosome / male germ cell nucleus /  chromosome segregation / chromosome segregation /  transcription coregulator binding / promoter-specific chromatin binding / sensory perception of sound / transcription coregulator binding / promoter-specific chromatin binding / sensory perception of sound /  brain development / response to radiation / brain development / response to radiation /  protein localization / protein localization /  動原体 / 動原体 /  認識 / DNA-binding transcription repressor activity, RNA polymerase II-specific / 認識 / DNA-binding transcription repressor activity, RNA polymerase II-specific /  nuclear matrix / nuclear matrix /  histone deacetylase binding / histone deacetylase binding /  紡錘体 / Activation of anterior HOX genes in hindbrain development during early embryogenesis / Separation of Sister Chromatids / transcription corepressor activity / double-strand break repair / 紡錘体 / Activation of anterior HOX genes in hindbrain development during early embryogenesis / Separation of Sister Chromatids / transcription corepressor activity / double-strand break repair /  染色体 / mitotic cell cycle / midbody / 染色体 / mitotic cell cycle / midbody /  double-stranded DNA binding double-stranded DNA binding類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.5 Å クライオ電子顕微鏡法 / 解像度: 6.5 Å | ||||||||||||
 データ登録者 データ登録者 | Shi, Z.B. / Bai, X.C. / Yu, H. | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: CTCF and R-loops are boundaries of cohesin-mediated DNA looping. 著者: Hongshan Zhang / Zhubing Shi / Edward J Banigan / Yoori Kim / Hongtao Yu / Xiao-Chen Bai / Ilya J Finkelstein /   要旨: Cohesin and CCCTC-binding factor (CTCF) are key regulatory proteins of three-dimensional (3D) genome organization. Cohesin extrudes DNA loops that are anchored by CTCF in a polar orientation. Here, ...Cohesin and CCCTC-binding factor (CTCF) are key regulatory proteins of three-dimensional (3D) genome organization. Cohesin extrudes DNA loops that are anchored by CTCF in a polar orientation. Here, we present direct evidence that CTCF binding polarity controls cohesin-mediated DNA looping. Using single-molecule imaging, we demonstrate that a critical N-terminal motif of CTCF blocks cohesin translocation and DNA looping. The cryo-EM structure of the cohesin-CTCF complex reveals that this CTCF motif ahead of zinc fingers can only reach its binding site on the STAG1 cohesin subunit when the N terminus of CTCF faces cohesin. Remarkably, a C-terminally oriented CTCF accelerates DNA compaction by cohesin. DNA-bound Cas9 and Cas12a ribonucleoproteins are also polar cohesin barriers, indicating that stalling may be intrinsic to cohesin itself. Finally, we show that RNA-DNA hybrids (R-loops) block cohesin-mediated DNA compaction in vitro and are enriched with cohesin subunits in vivo, likely forming TAD boundaries. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7w1m.cif.gz 7w1m.cif.gz | 837.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7w1m.ent.gz pdb7w1m.ent.gz | 631.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7w1m.json.gz 7w1m.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/w1/7w1m https://data.pdbj.org/pub/pdb/validation_reports/w1/7w1m ftp://data.pdbj.org/pub/pdb/validation_reports/w1/7w1m ftp://data.pdbj.org/pub/pdb/validation_reports/w1/7w1m | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  32252MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Structural maintenance of chromosomes protein ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 143485.094 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SMC1A, DXS423E, KIAA0178, SB1.8, SMC1, SMC1L1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SMC1A, DXS423E, KIAA0178, SB1.8, SMC1, SMC1L1 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q14683 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q14683 |
|---|---|
| #2: タンパク質 | 分子量: 141771.562 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SMC3, BAM, BMH, CSPG6, SMC3L1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SMC3, BAM, BMH, CSPG6, SMC3L1 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q9UQE7 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q9UQE7 |
-タンパク質 , 4種, 4分子 CDEH
| #3: タンパク質 | 分子量: 71556.102 Da / 分子数: 1 / Mutation: R172A, D279A, R450A / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: RAD21, HR21, KIAA0078, NXP1, SCC1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: RAD21, HR21, KIAA0078, NXP1, SCC1 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: O60216 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: O60216 |
|---|---|
| #4: タンパク質 |  コヒーシン / SCC3 homolog 1 / Stromal antigen 1 コヒーシン / SCC3 homolog 1 / Stromal antigen 1分子量: 144616.969 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: STAG1, SA1, SCC3 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: STAG1, SA1, SCC3 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q8WVM7 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q8WVM7 |
| #5: タンパク質 | 分子量: 167830.547 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: NIPBL, IDN3, SCC2 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: NIPBL, IDN3, SCC2 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q6KC79 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q6KC79 |
| #8: タンパク質 | 分子量: 82928.758 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: CTCF / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CTCF / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P49711 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P49711 |
-DNA鎖 , 2種, 2分子 FG
| #6: DNA鎖 | 分子量: 36202.281 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|---|
| #7: DNA鎖 | 分子量: 36605.387 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-非ポリマー , 3種, 15分子 




| #9: 化合物 |  アデノシン二リン酸 アデノシン二リン酸#10: 化合物 | #11: 化合物 | ChemComp-ZN / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) | ||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||
急速凍結 | 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 60 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2185704 | ||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 6.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 42704 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 236.05 Å2 | ||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj