+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7uzz | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
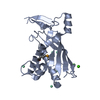
| タイトル | Staphylococcus epidermidis RP62a CRISPR tall effector complex | |||||||||||||||
 要素 要素 |
| |||||||||||||||
 キーワード キーワード | HYDROLASE/RNA / Type IIIA CRISPR / effector complex /  RNA binding protein (RNA結合タンパク質) / HYDROLASE-RNA complex RNA binding protein (RNA結合タンパク質) / HYDROLASE-RNA complex | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 exonuclease activity / exonuclease activity /  endonuclease activity / defense response to virus / endonuclease activity / defense response to virus /  RNA binding / RNA binding /  ATP binding ATP binding類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) | |||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.45 Å クライオ電子顕微鏡法 / 解像度: 4.45 Å | |||||||||||||||
 データ登録者 データ登録者 | Smith, E.M. / Ferrell, S.H. / Tokars, V.L. / Mondragon, A. | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Structures of an active type III-A CRISPR effector complex. 著者: Eric M Smith / Sé Ferrell / Valerie L Tokars / Alfonso Mondragón /  要旨: Clustered regularly interspaced short palindromic repeats (CRISPR) and their CRISPR-associated proteins (Cas) provide many prokaryotes with an adaptive immune system against invading genetic material. ...Clustered regularly interspaced short palindromic repeats (CRISPR) and their CRISPR-associated proteins (Cas) provide many prokaryotes with an adaptive immune system against invading genetic material. Type III CRISPR systems are unique in that they can degrade both RNA and DNA. In response to invading nucleic acids, they produce cyclic oligoadenylates that act as secondary messengers, activating cellular nucleases that aid in the immune response. Here, we present seven single-particle cryo-EM structures of the type III-A Staphylococcus epidermidis CRISPR effector complex. The structures reveal the intact S. epidermidis effector complex in an apo, ATP-bound, cognate target RNA-bound, and non-cognate target RNA-bound states and illustrate how the effector complex binds and presents crRNA. The complexes bound to target RNA capture the type III-A effector complex in a post-RNA cleavage state. The ATP-bound structures give details about how ATP binds to Cas10 to facilitate cyclic oligoadenylate production. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7uzz.cif.gz 7uzz.cif.gz | 438.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7uzz.ent.gz pdb7uzz.ent.gz | 360.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7uzz.json.gz 7uzz.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uz/7uzz https://data.pdbj.org/pub/pdb/validation_reports/uz/7uzz ftp://data.pdbj.org/pub/pdb/validation_reports/uz/7uzz ftp://data.pdbj.org/pub/pdb/validation_reports/uz/7uzz | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  26923MC  7uzwC  7uzxC  7uzyC  7v00C  7v01C  7v02C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-RNA鎖 , 1種, 1分子 G
| #1: RNA鎖 | 分子量: 11895.168 Da / 分子数: 1 断片: Staphylococcus epidermidis RP62A CRISPR RNA: Repeat plus Spacer sequence 1 由来タイプ: 天然 由来: (天然)   Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
|---|
-CRISPR system ... , 5種, 10分子 JIKEDCABHF
| #2: タンパク質 | 分子量: 15436.794 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)   Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A / 参照: UniProt: Q5HK90 #3: タンパク質 | | 分子量: 39449.125 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A / 参照: UniProt: Q5HK93 #4: タンパク質 | 分子量: 24033.975 Da / 分子数: 4 / 由来タイプ: 天然 由来: (天然)   Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A / 参照: UniProt: Q5HK91 #5: タンパク質 | | 分子量: 34551.938 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A / 参照: UniProt: Q5HK92 #6: タンパク質 | | 分子量: 87685.781 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A / 参照: UniProt: Q5HK89 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Staphylococcus epidermidis RP62a CRISPR tall effector complex タイプ: COMPLEX / Entity ID: all / 由来: NATURAL | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | ||||||||||||||||||||
| 由来(天然) | 生物種:   Staphylococcus epidermidis (表皮ブドウ球菌) Staphylococcus epidermidis (表皮ブドウ球菌)株: RP62A | ||||||||||||||||||||
| 緩衝液 | pH: 8 | ||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||
| 試料 | 濃度: 3.2 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||
| 試料支持 | 詳細: Grids were glow discharged for 10 seconds with 12 mA current (Pelco easiGlow) prior to use on Chameleon. グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Homemade | ||||||||||||||||||||
急速凍結 | 装置: SPOTITON / 凍結剤: ETHANE / 湿度: 95 % / 凍結前の試料温度: 277 K 詳細: Grids were prepared at the National Center for CryoEM Access and Training (NCCAT) using a Chameleon (SPT Labtech) blotless system. 300 mesh nanowire grids with multi-hole pattern carbon film ...詳細: Grids were prepared at the National Center for CryoEM Access and Training (NCCAT) using a Chameleon (SPT Labtech) blotless system. 300 mesh nanowire grids with multi-hole pattern carbon film and a thin layer of gold coating (~ 5 nm) (SPT Labtech)were used |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(補正後): 46772 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 800 nm / アライメント法: COMA FREE Bright-field microscopy / 倍率(補正後): 46772 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 800 nm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 61.07 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 6041 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1982259 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 4.45 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 101620 / アルゴリズム: FOURIER SPACE / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: THROUGHOUT | ||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 178.48 Å2 / Biso mean: 110.998 Å2 / Biso min: 30 Å2 | ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj