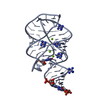
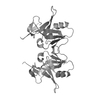
登録情報 データベース : PDB / ID : 7lqtタイトル Solution NMR structure of the PNUTS amino-terminal Domain fused to Myc Homology Box 0 Serine/threonine-protein phosphatase 1 regulatory subunit 10,Myc proto-oncogene protein fusion キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Rattus norvegicus (ドブネズミ)手法 / データ登録者 Lemak, A. / Wei, Y. / Duan, S. / Houliston, S. / Penn, L.Z. / Arrowsmith, C.H. ジャーナル : Nucleic Acids Res. / 年 : 2022タイトル : The MYC oncoprotein directly interacts with its chromatin cofactor PNUTS to recruit PP1 phosphatase.著者: Wei, Y. / Redel, C. / Ahlner, A. / Lemak, A. / Johansson-Akhe, I. / Houliston, S. / Kenney, T.M.G. / Tamachi, A. / Morad, V. / Duan, S. / Andrews, D.W. / Wallner, B. / Sunnerhagen, M. / ... 著者 : Wei, Y. / Redel, C. / Ahlner, A. / Lemak, A. / Johansson-Akhe, I. / Houliston, S. / Kenney, T.M.G. / Tamachi, A. / Morad, V. / Duan, S. / Andrews, D.W. / Wallner, B. / Sunnerhagen, M. / Arrowsmith, C.H. / Penn, L.Z. 履歴 登録 2021年2月15日 登録サイト / 処理サイト 改定 1.0 2021年3月3日 Provider / タイプ 改定 1.1 2022年3月23日 Group / カテゴリ / citation_author / database_2Item _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _database_2.pdbx_DOI / _database_2.pdbx_database_accession 改定 1.2 2022年4月20日 Group / カテゴリ / citation_authorItem _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID 改定 1.3 2023年6月14日 Group / カテゴリ / Item 改定 1.4 2024年5月15日 Group / Database references / カテゴリ / chem_comp_bond / database_2 / Item
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード NUCLEAR PROTEIN / PP1-PNUTS phosphatase complex / Regulatory Subunit / c-MYC oncoprotein / Myc Box 0
NUCLEAR PROTEIN / PP1-PNUTS phosphatase complex / Regulatory Subunit / c-MYC oncoprotein / Myc Box 0 機能・相同性情報
機能・相同性情報 排卵 / cellular response to xenobiotic stimulus => GO:0071466 / : / inner mitochondrial membrane organization / cellular response to interferon-alpha / : / flavonoid metabolic process / response to human chorionic gonadotropin / positive regulation of B cell apoptotic process / regulation of somatic stem cell population maintenance / PTW/PP1 phosphatase complex / cellular response to prolactin / cellular response to carbohydrate stimulus / Ub-specific processing proteases / cellular response to lectin / positive regulation of cellular respiration / response to xenobiotic stimulus => GO:0009410 / G0 to G1 transition / detection of mechanical stimulus involved in sensory perception of sound / negative regulation of cell division /
排卵 / cellular response to xenobiotic stimulus => GO:0071466 / : / inner mitochondrial membrane organization / cellular response to interferon-alpha / : / flavonoid metabolic process / response to human chorionic gonadotropin / positive regulation of B cell apoptotic process / regulation of somatic stem cell population maintenance / PTW/PP1 phosphatase complex / cellular response to prolactin / cellular response to carbohydrate stimulus / Ub-specific processing proteases / cellular response to lectin / positive regulation of cellular respiration / response to xenobiotic stimulus => GO:0009410 / G0 to G1 transition / detection of mechanical stimulus involved in sensory perception of sound / negative regulation of cell division /  protein phosphatase 1 binding / negative regulation of monocyte differentiation / cellular response to phorbol 13-acetate 12-myristate / cellular response to hydrostatic pressure /
protein phosphatase 1 binding / negative regulation of monocyte differentiation / cellular response to phorbol 13-acetate 12-myristate / cellular response to hydrostatic pressure /  regulation of oxidative phosphorylation / positive regulation of telomere maintenance / B cell apoptotic process / cellular response to testosterone stimulus / positive regulation of oxidative phosphorylation / negative regulation of glucose import / response to alkaloid / cellular response to fibroblast growth factor stimulus / amino acid transport / middle ear morphogenesis / cellular response to angiotensin / positive regulation of mitochondrial membrane potential /
regulation of oxidative phosphorylation / positive regulation of telomere maintenance / B cell apoptotic process / cellular response to testosterone stimulus / positive regulation of oxidative phosphorylation / negative regulation of glucose import / response to alkaloid / cellular response to fibroblast growth factor stimulus / amino acid transport / middle ear morphogenesis / cellular response to angiotensin / positive regulation of mitochondrial membrane potential /  regulation of telomere maintenance /
regulation of telomere maintenance /  skeletal system morphogenesis / negative regulation of cardiac muscle cell apoptotic process /
skeletal system morphogenesis / negative regulation of cardiac muscle cell apoptotic process /  顔料 / protein-DNA complex disassembly / positive regulation of mesenchymal cell proliferation / protein phosphatase inhibitor activity / positive regulation of DNA biosynthetic process / branching involved in ureteric bud morphogenesis / hypothalamus development / cellular response to platelet-derived growth factor stimulus / positive regulation of catalytic activity / positive regulation of ATP biosynthetic process / positive regulation of smooth muscle cell migration / transformation of host cell by virus /
顔料 / protein-DNA complex disassembly / positive regulation of mesenchymal cell proliferation / protein phosphatase inhibitor activity / positive regulation of DNA biosynthetic process / branching involved in ureteric bud morphogenesis / hypothalamus development / cellular response to platelet-derived growth factor stimulus / positive regulation of catalytic activity / positive regulation of ATP biosynthetic process / positive regulation of smooth muscle cell migration / transformation of host cell by virus /  E-box binding / skeletal muscle cell differentiation / cellular response to cytokine stimulus / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process / cellular response to organic cyclic compound / chromosome organization / positive regulation of transcription initiation by RNA polymerase II / positive regulation of DNA binding / cellular response to interleukin-1 / cellular response to estrogen stimulus / cis-regulatory region sequence-specific DNA binding / positive regulation of cell cycle / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cellular response to retinoic acid / ovarian follicle development / positive regulation of glial cell proliferation / positive regulation of telomerase activity / cellular response to epidermal growth factor stimulus / regulation of mitotic cell cycle / ERK1 and ERK2 cascade / positive regulation of glycolytic process / negative regulation of protein binding / DNA-templated transcription initiation / positive regulation of apoptotic signaling pathway / positive regulation of epithelial cell proliferation /
E-box binding / skeletal muscle cell differentiation / cellular response to cytokine stimulus / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process / cellular response to organic cyclic compound / chromosome organization / positive regulation of transcription initiation by RNA polymerase II / positive regulation of DNA binding / cellular response to interleukin-1 / cellular response to estrogen stimulus / cis-regulatory region sequence-specific DNA binding / positive regulation of cell cycle / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cellular response to retinoic acid / ovarian follicle development / positive regulation of glial cell proliferation / positive regulation of telomerase activity / cellular response to epidermal growth factor stimulus / regulation of mitotic cell cycle / ERK1 and ERK2 cascade / positive regulation of glycolytic process / negative regulation of protein binding / DNA-templated transcription initiation / positive regulation of apoptotic signaling pathway / positive regulation of epithelial cell proliferation /  liver regeneration / response to gamma radiation / positive regulation of smooth muscle cell proliferation /
liver regeneration / response to gamma radiation / positive regulation of smooth muscle cell proliferation /  ユークロマチン / response to radiation / protein processing
ユークロマチン / response to radiation / protein processing
 Rattus norvegicus (ドブネズミ)
Rattus norvegicus (ドブネズミ) 溶液NMR /
溶液NMR /  molecular dynamics
molecular dynamics  データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2022
ジャーナル: Nucleic Acids Res. / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7lqt.cif.gz
7lqt.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7lqt.ent.gz
pdb7lqt.ent.gz PDB形式
PDB形式 7lqt.json.gz
7lqt.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/lq/7lqt
https://data.pdbj.org/pub/pdb/validation_reports/lq/7lqt ftp://data.pdbj.org/pub/pdb/validation_reports/lq/7lqt
ftp://data.pdbj.org/pub/pdb/validation_reports/lq/7lqt
 リンク
リンク 集合体
集合体
 要素
要素
 Rattus norvegicus (ドブネズミ) / 遺伝子: Ppp1r10, Cat53, Pnuts, Myc / 発現宿主:
Rattus norvegicus (ドブネズミ) / 遺伝子: Ppp1r10, Cat53, Pnuts, Myc / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: O55000, UniProt: P09416
Escherichia coli (大腸菌) / 参照: UniProt: O55000, UniProt: P09416 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 molecular dynamics / ソフトェア番号: 1
molecular dynamics / ソフトェア番号: 1  ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj





