+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6ges | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of ERK1 covalently bound to SM1-71 | ||||||
 要素 要素 | Mitogen-activated protein kinase 3 | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  kinase (キナーゼ) / covalent inhibitor / MAPK / kinase (キナーゼ) / covalent inhibitor / MAPK /  Structural Genomics (構造ゲノミクス) / Structural Genomics (構造ゲノミクス) /  Structural Genomics Consortium / SGC Structural Genomics Consortium / SGC | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of xenophagy /  xenophagy / RAF-independent MAPK1/3 activation / Suppression of apoptosis / Signaling by Activin / Gastrin-CREB signalling pathway via PKC and MAPK / cardiac neural crest cell development involved in heart development / caveolin-mediated endocytosis / ERKs are inactivated / response to epidermal growth factor ...positive regulation of xenophagy / xenophagy / RAF-independent MAPK1/3 activation / Suppression of apoptosis / Signaling by Activin / Gastrin-CREB signalling pathway via PKC and MAPK / cardiac neural crest cell development involved in heart development / caveolin-mediated endocytosis / ERKs are inactivated / response to epidermal growth factor ...positive regulation of xenophagy /  xenophagy / RAF-independent MAPK1/3 activation / Suppression of apoptosis / Signaling by Activin / Gastrin-CREB signalling pathway via PKC and MAPK / cardiac neural crest cell development involved in heart development / caveolin-mediated endocytosis / ERKs are inactivated / response to epidermal growth factor / Signaling by MAP2K mutants / Signaling by NODAL / RSK activation / Golgi Cisternae Pericentriolar Stack Reorganization / regulation of cellular pH / positive regulation of macrophage proliferation / positive regulation of cyclase activity / outer ear morphogenesis / Regulation of the apoptosome activity / cartilage development / regulation of Golgi inheritance / interleukin-1-mediated signaling pathway / trachea formation / Negative feedback regulation of MAPK pathway / regulation of early endosome to late endosome transport / regulation of stress-activated MAPK cascade / IFNG signaling activates MAPKs / Frs2-mediated activation / positive regulation of macrophage chemotaxis / lung morphogenesis / ERBB2-ERBB3 signaling pathway / response to exogenous dsRNA / regulation of cytoskeleton organization / Activation of the AP-1 family of transcription factors / face development / MAPK3 (ERK1) activation / ERK/MAPK targets / xenophagy / RAF-independent MAPK1/3 activation / Suppression of apoptosis / Signaling by Activin / Gastrin-CREB signalling pathway via PKC and MAPK / cardiac neural crest cell development involved in heart development / caveolin-mediated endocytosis / ERKs are inactivated / response to epidermal growth factor / Signaling by MAP2K mutants / Signaling by NODAL / RSK activation / Golgi Cisternae Pericentriolar Stack Reorganization / regulation of cellular pH / positive regulation of macrophage proliferation / positive regulation of cyclase activity / outer ear morphogenesis / Regulation of the apoptosome activity / cartilage development / regulation of Golgi inheritance / interleukin-1-mediated signaling pathway / trachea formation / Negative feedback regulation of MAPK pathway / regulation of early endosome to late endosome transport / regulation of stress-activated MAPK cascade / IFNG signaling activates MAPKs / Frs2-mediated activation / positive regulation of macrophage chemotaxis / lung morphogenesis / ERBB2-ERBB3 signaling pathway / response to exogenous dsRNA / regulation of cytoskeleton organization / Activation of the AP-1 family of transcription factors / face development / MAPK3 (ERK1) activation / ERK/MAPK targets /  仮足 / RUNX2 regulates osteoblast differentiation / Bergmann glial cell differentiation / positive regulation of telomere capping / thyroid gland development / Advanced glycosylation endproduct receptor signaling / RHO GTPases Activate NADPH Oxidases / Estrogen-dependent nuclear events downstream of ESR-membrane signaling / Regulation of HSF1-mediated heat shock response / 仮足 / RUNX2 regulates osteoblast differentiation / Bergmann glial cell differentiation / positive regulation of telomere capping / thyroid gland development / Advanced glycosylation endproduct receptor signaling / RHO GTPases Activate NADPH Oxidases / Estrogen-dependent nuclear events downstream of ESR-membrane signaling / Regulation of HSF1-mediated heat shock response /  MAP kinase activity / MAP kinase activity /  regulation of ossification / RHO GTPases Activate WASPs and WAVEs / regulation of ossification / RHO GTPases Activate WASPs and WAVEs /  分裂促進因子活性化タンパク質キナーゼ / 分裂促進因子活性化タンパク質キナーゼ /  phosphatase binding / phosphatase binding /  signal transduction in response to DNA damage / Nuclear events stimulated by ALK signaling in cancer / Signal attenuation / BMP signaling pathway / Schwann cell development / stress-activated MAPK cascade / Growth hormone receptor signaling / peptidyl-tyrosine autophosphorylation / lipopolysaccharide-mediated signaling pathway / positive regulation of telomerase activity / sensory perception of pain / negative regulation of TORC1 signaling / cellular response to cadmium ion / positive regulation of telomere maintenance via telomerase / ERK1 and ERK2 cascade / NPAS4 regulates expression of target genes / cellular response to amino acid starvation / signal transduction in response to DNA damage / Nuclear events stimulated by ALK signaling in cancer / Signal attenuation / BMP signaling pathway / Schwann cell development / stress-activated MAPK cascade / Growth hormone receptor signaling / peptidyl-tyrosine autophosphorylation / lipopolysaccharide-mediated signaling pathway / positive regulation of telomerase activity / sensory perception of pain / negative regulation of TORC1 signaling / cellular response to cadmium ion / positive regulation of telomere maintenance via telomerase / ERK1 and ERK2 cascade / NPAS4 regulates expression of target genes / cellular response to amino acid starvation /  髄鞘 / RNA Polymerase I Promoter Opening / NCAM signaling for neurite out-growth / phosphotyrosine residue binding / ESR-mediated signaling / insulin-like growth factor receptor signaling pathway / thymus development / Regulation of PTEN gene transcription / Signal transduction by L1 / 髄鞘 / RNA Polymerase I Promoter Opening / NCAM signaling for neurite out-growth / phosphotyrosine residue binding / ESR-mediated signaling / insulin-like growth factor receptor signaling pathway / thymus development / Regulation of PTEN gene transcription / Signal transduction by L1 /  カベオラ / Negative regulation of FGFR3 signaling / Downregulation of SMAD2/3:SMAD4 transcriptional activity / FCERI mediated MAPK activation / Negative regulation of FGFR2 signaling / FCGR3A-mediated phagocytosis / Negative regulation of FGFR4 signaling / Negative regulation of FGFR1 signaling / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription / Spry regulation of FGF signaling / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / modulation of chemical synaptic transmission / Oncogene Induced Senescence / Regulation of actin dynamics for phagocytic cup formation / ISG15 antiviral mechanism / cellular response to reactive oxygen species / cellular response to mechanical stimulus / Negative regulation of MAPK pathway / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants カベオラ / Negative regulation of FGFR3 signaling / Downregulation of SMAD2/3:SMAD4 transcriptional activity / FCERI mediated MAPK activation / Negative regulation of FGFR2 signaling / FCGR3A-mediated phagocytosis / Negative regulation of FGFR4 signaling / Negative regulation of FGFR1 signaling / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription / Spry regulation of FGF signaling / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / modulation of chemical synaptic transmission / Oncogene Induced Senescence / Regulation of actin dynamics for phagocytic cup formation / ISG15 antiviral mechanism / cellular response to reactive oxygen species / cellular response to mechanical stimulus / Negative regulation of MAPK pathway / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.07 Å 分子置換 / 解像度: 2.07 Å | ||||||
 データ登録者 データ登録者 | Chaikuad, A. / Suman, R. / Arrowsmith, C.H. / Edwards, A.M. / Bountra, C. / Gray, N.S. / Knapp, S. / Structural Genomics Consortium (SGC) | ||||||
 引用 引用 |  ジャーナル: Cell Chem Biol / 年: 2019 ジャーナル: Cell Chem Biol / 年: 2019タイトル: Leveraging Compound Promiscuity to Identify Targetable Cysteines within the Kinome. 著者: Rao, S. / Gurbani, D. / Du, G. / Everley, R.A. / Browne, C.M. / Chaikuad, A. / Tan, L. / Schroder, M. / Gondi, S. / Ficarro, S.B. / Sim, T. / Kim, N.D. / Berberich, M.J. / Knapp, S. / Marto, ...著者: Rao, S. / Gurbani, D. / Du, G. / Everley, R.A. / Browne, C.M. / Chaikuad, A. / Tan, L. / Schroder, M. / Gondi, S. / Ficarro, S.B. / Sim, T. / Kim, N.D. / Berberich, M.J. / Knapp, S. / Marto, J.A. / Westover, K.D. / Sorger, P.K. / Gray, N.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6ges.cif.gz 6ges.cif.gz | 300.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6ges.ent.gz pdb6ges.ent.gz | 242.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6ges.json.gz 6ges.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ge/6ges https://data.pdbj.org/pub/pdb/validation_reports/ge/6ges ftp://data.pdbj.org/pub/pdb/validation_reports/ge/6ges ftp://data.pdbj.org/pub/pdb/validation_reports/ge/6ges | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||
| 2 | 
| ||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 0 / Ens-ID: 1 / Beg auth comp-ID: PRO / Beg label comp-ID: PRO / End auth comp-ID: GLY / End label comp-ID: GLY / Refine code: 0 / Auth seq-ID: 25 - 374 / Label seq-ID: 26 - 375
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 43277.566 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: MAPK3, ERK1, PRKM3 / プラスミド: pNIC28-Bsa4 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MAPK3, ERK1, PRKM3 / プラスミド: pNIC28-Bsa4 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): R3-pRARE2 Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): R3-pRARE2参照: UniProt: P27361,  分裂促進因子活性化タンパク質キナーゼ 分裂促進因子活性化タンパク質キナーゼ |
|---|
-非ポリマー , 5種, 322分子 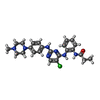
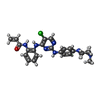







| #2: 化合物 | | #3: 化合物 | ChemComp-EWH / ~{ | #4: 化合物 |  硫酸塩 硫酸塩#5: 化合物 | ChemComp-EDO /  エチレングリコール エチレングリコール#6: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.18 Å3/Da / 溶媒含有率: 43.69 % |
|---|---|
結晶化 | 温度: 277.14 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 / 詳細: 34% PEG 4k, 0.2M Li2SO4, 0.1M tris 7.5 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06SA / 波長: 0.99986 Å / ビームライン: X06SA / 波長: 0.99986 Å |
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2018年4月18日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.99986 Å / 相対比: 1 : 0.99986 Å / 相対比: 1 |
| 反射 | 解像度: 2.07→47.02 Å / Num. obs: 44884 / % possible obs: 98.9 % / 冗長度: 5.4 % / Biso Wilson estimate: 35.9 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.061 / Net I/σ(I): 13.5 |
| 反射 シェル | 解像度: 2.07→2.13 Å / 冗長度: 5.4 % / Rmerge(I) obs: 0.786 / Mean I/σ(I) obs: 2 / Num. unique obs: 3496 / CC1/2: 0.799 / % possible all: 99.3 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 4qtb 解像度: 2.07→47.02 Å / Cor.coef. Fo:Fc: 0.971 / Cor.coef. Fo:Fc free: 0.951 / SU B: 10.836 / SU ML: 0.142 / 交差検証法: THROUGHOUT / ESU R: 0.209 / ESU R Free: 0.171 / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 45.749 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.07→47.02 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj
































