+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6et4 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | HUMAN DIHYDROOROTATE DEHYDROGENASE IN COMPLEX WITH NOVEL INHIBITOR | ||||||
 要素 要素 | Dihydroorotate dehydrogenase (quinone), mitochondrial ジヒドロオロト酸デヒドロゲナーゼ (キノン) ジヒドロオロト酸デヒドロゲナーゼ (キノン) | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  DHODH / DHODH /  CANCER (悪性腫瘍) / P53 ACTIVATION / PYRIMIDINE SYNTHESIS CANCER (悪性腫瘍) / P53 ACTIVATION / PYRIMIDINE SYNTHESIS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pyrimidine ribonucleotide biosynthetic process /  dihydroorotate dehydrogenase (quinone) activity / dihydroorotate dehydrogenase (quinone) activity /  ジヒドロオロト酸デヒドロゲナーゼ (キノン) / ジヒドロオロト酸デヒドロゲナーゼ (キノン) /  dihydroorotate dehydrogenase activity / dihydroorotate dehydrogenase activity /  dihydroorotase activity / dihydroorotase activity /  Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process / Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process /  ミトコンドリア内膜 ...pyrimidine ribonucleotide biosynthetic process / ミトコンドリア内膜 ...pyrimidine ribonucleotide biosynthetic process /  dihydroorotate dehydrogenase (quinone) activity / dihydroorotate dehydrogenase (quinone) activity /  ジヒドロオロト酸デヒドロゲナーゼ (キノン) / ジヒドロオロト酸デヒドロゲナーゼ (キノン) /  dihydroorotate dehydrogenase activity / dihydroorotate dehydrogenase activity /  dihydroorotase activity / dihydroorotase activity /  Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process / Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process /  ミトコンドリア内膜 / ミトコンドリア内膜 /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å | ||||||
 データ登録者 データ登録者 | Hakansson, M. / Walse, B. / Gustavsson, A.-L. / Lain, S. | ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: A DHODH inhibitor increases p53 synthesis and enhances tumor cell killing by p53 degradation blockage. 著者: Ladds, M.J.G.W. / van Leeuwen, I.M.M. / Drummond, C.J. / Chu, S. / Healy, A.R. / Popova, G. / Pastor Fernandez, A. / Mollick, T. / Darekar, S. / Sedimbi, S.K. / Nekulova, M. / Sachweh, M.C.C. ...著者: Ladds, M.J.G.W. / van Leeuwen, I.M.M. / Drummond, C.J. / Chu, S. / Healy, A.R. / Popova, G. / Pastor Fernandez, A. / Mollick, T. / Darekar, S. / Sedimbi, S.K. / Nekulova, M. / Sachweh, M.C.C. / Campbell, J. / Higgins, M. / Tuck, C. / Popa, M. / Safont, M.M. / Gelebart, P. / Fandalyuk, Z. / Thompson, A.M. / Svensson, R. / Gustavsson, A.L. / Johansson, L. / Farnegardh, K. / Yngve, U. / Saleh, A. / Haraldsson, M. / D'Hollander, A.C.A. / Franco, M. / Zhao, Y. / Hakansson, M. / Walse, B. / Larsson, K. / Peat, E.M. / Pelechano, V. / Lunec, J. / Vojtesek, B. / Carmena, M. / Earnshaw, W.C. / McCarthy, A.R. / Westwood, N.J. / Arsenian-Henriksson, M. / Lane, D.P. / Bhatia, R. / McCormack, E. / Lain, S. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "AB" IN EACH CHAIN ON SHEET RECORDS BELOW ...SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "AB" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 8-STRANDED BARREL THIS IS REPRESENTED BY A 9-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6et4.cif.gz 6et4.cif.gz | 184.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6et4.ent.gz pdb6et4.ent.gz | 143 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6et4.json.gz 6et4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/et/6et4 https://data.pdbj.org/pub/pdb/validation_reports/et/6et4 ftp://data.pdbj.org/pub/pdb/validation_reports/et/6et4 ftp://data.pdbj.org/pub/pdb/validation_reports/et/6et4 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  4oqvS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  ジヒドロオロト酸デヒドロゲナーゼ (キノン) / DHOdehase / Dihydroorotate oxidase ジヒドロオロト酸デヒドロゲナーゼ (キノン) / DHOdehase / Dihydroorotate oxidase分子量: 39856.535 Da / 分子数: 1 / 断片: N-TERMINALLY TRUNCATED HUMAN DHODH, RESIDUES 29-395 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / Cell: U937 / 遺伝子: DHODH / 発現宿主: Homo sapiens (ヒト) / Cell: U937 / 遺伝子: DHODH / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌)参照: UniProt: Q02127,  ジヒドロオロト酸デヒドロゲナーゼ (キノン) ジヒドロオロト酸デヒドロゲナーゼ (キノン) |
|---|
-非ポリマー , 9種, 273分子 
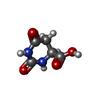














| #2: 化合物 | ChemComp-FMN /  フラビンモノヌクレオチド フラビンモノヌクレオチド | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| #3: 化合物 | ChemComp-DOR / ( | ||||||||
| #4: 化合物 | ChemComp-SDV / | ||||||||
| #5: 化合物 | ChemComp-DDQ / | ||||||||
| #6: 化合物 | ChemComp-ACY /  酢酸 酢酸#7: 化合物 |  グリセリン グリセリン#8: 化合物 |  硫酸塩 硫酸塩#9: 化合物 |  塩化物 塩化物#10: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 配列の詳細 | START AT POSITION 31 IN SEQUENCE TGDER.. ETC (FIRST TWO AA MA HAS BEEN ADDED) |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.67 Å3/Da / 溶媒含有率: 66.47 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.8 詳細: 30% glycerol, 100 mM Sodium acetate pH 4.8, 2 M Ammoniumsulphate PH範囲: 4.8 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I02 / 波長: 0.9795 Å / ビームライン: I02 / 波長: 0.9795 Å |
| 検出器 | タイプ: DECTRIS PILATUS3 6M / 検出器: PIXEL / 日付: 2015年8月6日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9795 Å / 相対比: 1 : 0.9795 Å / 相対比: 1 |
| 反射 | 解像度: 1.7→30 Å / Num. obs: 64865 / % possible obs: 99.1 % / Observed criterion σ(I): 0 / 冗長度: 10.7 % / Rmerge(I) obs: 0.06 / Net I/σ(I): 23.4 |
| 反射 シェル | 解像度: 1.7→1.74 Å / 冗長度: 7.6 % / Rmerge(I) obs: 0.46 / Mean I/σ(I) obs: 4.3 / % possible all: 86.1 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 4OQV 解像度: 1.7→30 Å / Cor.coef. Fo:Fc: 0.98 / Cor.coef. Fo:Fc free: 0.969 / SU B: 1.863 / SU ML: 0.028 / 交差検証法: THROUGHOUT / ESU R: 0.059 / ESU R Free: 0.056 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. U VALUES REFINED INDIVIDUALLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 26.525 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→30 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj










