| 登録情報 | データベース: PDB / ID: 5zaz
|
|---|
| タイトル | Solution structure of integrin b2 monomer tranmembrane domain in bicelle |
|---|
 要素 要素 | Integrin beta-2 |
|---|
 キーワード キーワード |  CELL ADHESION (細胞接着) / integrin b2 / CELL ADHESION (細胞接着) / integrin b2 /  protein-lipid interaction / protein-lipid interaction /  phospholipids (リン脂質) / phospholipids (リン脂質) /  Ca2+ Ca2+ |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
integrin alphaX-beta2 complex / integrin alphaM-beta2 complex / positive regulation of prostaglandin-E synthase activity / positive regulation of neutrophil degranulation / integrin alphaL-beta2 complex / ICAM-3 receptor activity / complement component C3b binding / neutrophil migration / Toll Like Receptor 4 (TLR4) Cascade / negative regulation of dopamine metabolic process ...integrin alphaX-beta2 complex / integrin alphaM-beta2 complex / positive regulation of prostaglandin-E synthase activity / positive regulation of neutrophil degranulation / integrin alphaL-beta2 complex / ICAM-3 receptor activity / complement component C3b binding / neutrophil migration / Toll Like Receptor 4 (TLR4) Cascade / negative regulation of dopamine metabolic process / cell-cell adhesion via plasma-membrane adhesion molecules / heterotypic cell-cell adhesion /  integrin complex / positive regulation of leukocyte adhesion to vascular endothelial cell / integrin complex / positive regulation of leukocyte adhesion to vascular endothelial cell /  phagocytosis, engulfment / leukocyte cell-cell adhesion / receptor clustering / amyloid-beta clearance / endodermal cell differentiation / plasma membrane raft / tertiary granule membrane / ficolin-1-rich granule membrane / cellular response to low-density lipoprotein particle stimulus / positive regulation of protein targeting to membrane / Integrin cell surface interactions / specific granule membrane / regulation of peptidyl-tyrosine phosphorylation / phagocytosis, engulfment / leukocyte cell-cell adhesion / receptor clustering / amyloid-beta clearance / endodermal cell differentiation / plasma membrane raft / tertiary granule membrane / ficolin-1-rich granule membrane / cellular response to low-density lipoprotein particle stimulus / positive regulation of protein targeting to membrane / Integrin cell surface interactions / specific granule membrane / regulation of peptidyl-tyrosine phosphorylation /  heat shock protein binding / heat shock protein binding /  cell adhesion molecule binding / cell adhesion molecule binding /  receptor-mediated endocytosis / cell-matrix adhesion / receptor-mediated endocytosis / cell-matrix adhesion /  好中球 / positive regulation of superoxide anion generation / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall / microglial cell activation / 好中球 / positive regulation of superoxide anion generation / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall / microglial cell activation /  receptor internalization / receptor internalization /  細胞接着 / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / 細胞接着 / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell /  extracellular vesicle / extracellular vesicle /  integrin binding / cell-cell signaling / integrin binding / cell-cell signaling /  amyloid-beta binding / regulation of cell shape / Interleukin-4 and Interleukin-13 signaling / amyloid-beta binding / regulation of cell shape / Interleukin-4 and Interleukin-13 signaling /  receptor complex / receptor complex /  細胞接着 / 細胞接着 /  炎症 / external side of plasma membrane / 炎症 / external side of plasma membrane /  focal adhesion / apoptotic process / Neutrophil degranulation / focal adhesion / apoptotic process / Neutrophil degranulation /  protein kinase binding / protein kinase binding /  細胞膜 / extracellular exosome / 細胞膜 / extracellular exosome /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞膜類似検索 - 分子機能 細胞膜類似検索 - 分子機能 Integrin beta-2 subunit /  Cytochrome c1, transmembrane anchor, C-terminal / Integrin beta subunit, cytoplasmic domain / Integrin beta cytoplasmic domain / Integrin_b_cyt / Integrin beta tail domain / Integrin beta subunit, tail / Integrin beta tail domain superfamily / Integrin_B_tail / Integrin beta subunit, VWA domain ...Integrin beta-2 subunit / Cytochrome c1, transmembrane anchor, C-terminal / Integrin beta subunit, cytoplasmic domain / Integrin beta cytoplasmic domain / Integrin_b_cyt / Integrin beta tail domain / Integrin beta subunit, tail / Integrin beta tail domain superfamily / Integrin_B_tail / Integrin beta subunit, VWA domain ...Integrin beta-2 subunit /  Cytochrome c1, transmembrane anchor, C-terminal / Integrin beta subunit, cytoplasmic domain / Integrin beta cytoplasmic domain / Integrin_b_cyt / Integrin beta tail domain / Integrin beta subunit, tail / Integrin beta tail domain superfamily / Integrin_B_tail / Integrin beta subunit, VWA domain / Integrin beta subunit / Integrin beta N-terminal / Integrin beta chain VWA domain / Integrin plexin domain / Integrins beta chain cysteine-rich domain signature. / Integrin beta subunits (N-terminal portion of extracellular region) / Integrin domain superfamily / PSI domain / domain found in Plexins, Semaphorins and Integrins / von Willebrand factor A-like domain superfamily / EGF-like domain signature 2. / EGF-like domain signature 1. / Single alpha-helices involved in coiled-coils or other helix-helix interfaces / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 Cytochrome c1, transmembrane anchor, C-terminal / Integrin beta subunit, cytoplasmic domain / Integrin beta cytoplasmic domain / Integrin_b_cyt / Integrin beta tail domain / Integrin beta subunit, tail / Integrin beta tail domain superfamily / Integrin_B_tail / Integrin beta subunit, VWA domain / Integrin beta subunit / Integrin beta N-terminal / Integrin beta chain VWA domain / Integrin plexin domain / Integrins beta chain cysteine-rich domain signature. / Integrin beta subunits (N-terminal portion of extracellular region) / Integrin domain superfamily / PSI domain / domain found in Plexins, Semaphorins and Integrins / von Willebrand factor A-like domain superfamily / EGF-like domain signature 2. / EGF-like domain signature 1. / Single alpha-helices involved in coiled-coils or other helix-helix interfaces / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / torsion angle dynamics 溶液NMR / torsion angle dynamics |
|---|
 データ登録者 データ登録者 | Li, H. / Guo, J. / Xu, C. |
|---|
| 資金援助 |  中国, 12件 中国, 12件 | 組織 | 認可番号 | 国 |
|---|
| NSFC | 31470734 |  中国 中国 | | NSFC | 31670751 |  中国 中国 | | MOST | 2014CB541903 |  中国 中国 | | CAS Key Research Program of Frontier Sciences | QYZDB-SSW-SMC048 |  中国 中国 | | CAS Facility-based Open Research Program | |  中国 中国 | | NSFC Innovative Research Group | 31621003 |  中国 中国 | | NSFC Key Program | 31530022 |  中国 中国 | | Ten Thousand Talent Program National Program for Support of Top-notch Young Professionals of China | |  中国 中国 | | STSMC Key Program | 16JC1404800 |  中国 中国 | | NSFC National Science Fund for Distinguished Young Scholars | 31425009 |  中国 中国 | | STSMC Outstanding Academic Leader Program | 15XD1504200 |  中国 中国 | | CAS Strategic Priority Research Program | XDB08020100 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: PLoS Biol. / 年: 2018 ジャーナル: PLoS Biol. / 年: 2018
タイトル: Intramembrane ionic protein-lipid interaction regulates integrin structure and function.
著者: Guo, J. / Zhang, Y. / Li, H. / Chu, H. / Wang, Q. / Jiang, S. / Li, Y. / Shen, H. / Li, G. / Chen, J. / Xu, C. |
|---|
| 履歴 | | 登録 | 2018年2月9日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2018年10月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年11月28日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Data collection / Database references / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_nmr_software / pdbx_nmr_spectrometer
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model |
|---|
| 改定 1.3 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード CELL ADHESION (細胞接着) / integrin b2 /
CELL ADHESION (細胞接着) / integrin b2 /  protein-lipid interaction /
protein-lipid interaction /  phospholipids (リン脂質) /
phospholipids (リン脂質) /  Ca2+
Ca2+ 機能・相同性情報
機能・相同性情報 integrin complex / positive regulation of leukocyte adhesion to vascular endothelial cell /
integrin complex / positive regulation of leukocyte adhesion to vascular endothelial cell /  phagocytosis, engulfment / leukocyte cell-cell adhesion / receptor clustering / amyloid-beta clearance / endodermal cell differentiation / plasma membrane raft / tertiary granule membrane / ficolin-1-rich granule membrane / cellular response to low-density lipoprotein particle stimulus / positive regulation of protein targeting to membrane / Integrin cell surface interactions / specific granule membrane / regulation of peptidyl-tyrosine phosphorylation /
phagocytosis, engulfment / leukocyte cell-cell adhesion / receptor clustering / amyloid-beta clearance / endodermal cell differentiation / plasma membrane raft / tertiary granule membrane / ficolin-1-rich granule membrane / cellular response to low-density lipoprotein particle stimulus / positive regulation of protein targeting to membrane / Integrin cell surface interactions / specific granule membrane / regulation of peptidyl-tyrosine phosphorylation /  heat shock protein binding /
heat shock protein binding /  cell adhesion molecule binding /
cell adhesion molecule binding /  receptor-mediated endocytosis / cell-matrix adhesion /
receptor-mediated endocytosis / cell-matrix adhesion /  好中球 / positive regulation of superoxide anion generation / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall / microglial cell activation /
好中球 / positive regulation of superoxide anion generation / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall / microglial cell activation /  receptor internalization /
receptor internalization /  細胞接着 / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell /
細胞接着 / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell /  extracellular vesicle /
extracellular vesicle /  integrin binding / cell-cell signaling /
integrin binding / cell-cell signaling /  amyloid-beta binding / regulation of cell shape / Interleukin-4 and Interleukin-13 signaling /
amyloid-beta binding / regulation of cell shape / Interleukin-4 and Interleukin-13 signaling /  receptor complex /
receptor complex /  細胞接着 /
細胞接着 /  炎症 / external side of plasma membrane /
炎症 / external side of plasma membrane /  focal adhesion / apoptotic process / Neutrophil degranulation /
focal adhesion / apoptotic process / Neutrophil degranulation /  protein kinase binding /
protein kinase binding /  細胞膜 / extracellular exosome /
細胞膜 / extracellular exosome /  生体膜 /
生体膜 /  metal ion binding /
metal ion binding /  細胞膜
細胞膜
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR / torsion angle dynamics
溶液NMR / torsion angle dynamics  データ登録者
データ登録者 中国, 12件
中国, 12件  引用
引用 ジャーナル: PLoS Biol. / 年: 2018
ジャーナル: PLoS Biol. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5zaz.cif.gz
5zaz.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5zaz.ent.gz
pdb5zaz.ent.gz PDB形式
PDB形式 5zaz.json.gz
5zaz.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/za/5zaz
https://data.pdbj.org/pub/pdb/validation_reports/za/5zaz ftp://data.pdbj.org/pub/pdb/validation_reports/za/5zaz
ftp://data.pdbj.org/pub/pdb/validation_reports/za/5zaz リンク
リンク 集合体
集合体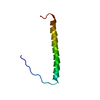
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: ITGB2, CD18, MFI7 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ITGB2, CD18, MFI7 / 発現宿主: 
 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P05107
Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P05107 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj






