+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5xng | ||||||
|---|---|---|---|---|---|---|---|
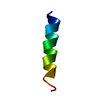
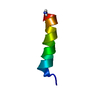
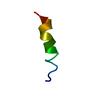
| タイトル | EFK17A structure in Microgel MAA60 | ||||||
 要素 要素 | Cathelicidin antimicrobial peptide | ||||||
 キーワード キーワード |  DE NOVO PROTEIN (De novo) / DE NOVO PROTEIN (De novo) /  ANTIMICROBIAL PROTEIN (抗微生物ペプチド) / ANTIMICROBIAL PROTEIN (抗微生物ペプチド) /  antimicrobial peptide (抗微生物ペプチド) / helical structure / peptide derived from human LL37 antimicrobial peptide (抗微生物ペプチド) / helical structure / peptide derived from human LL37 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 cytolysis / killing by host of symbiont cells / cytolysis / killing by host of symbiont cells /  好中球 / 好中球 /  specific granule / cellular response to peptidoglycan / cellular response to interleukin-6 / specific granule / cellular response to peptidoglycan / cellular response to interleukin-6 /  抗微生物ペプチド / cellular response to interleukin-1 / 抗微生物ペプチド / cellular response to interleukin-1 /  innate immune response in mucosa / cell projection ... innate immune response in mucosa / cell projection ... cytolysis / killing by host of symbiont cells / cytolysis / killing by host of symbiont cells /  好中球 / 好中球 /  specific granule / cellular response to peptidoglycan / cellular response to interleukin-6 / specific granule / cellular response to peptidoglycan / cellular response to interleukin-6 /  抗微生物ペプチド / cellular response to interleukin-1 / 抗微生物ペプチド / cellular response to interleukin-1 /  innate immune response in mucosa / cell projection / innate immune response in mucosa / cell projection /  lipopolysaccharide binding / specific granule lumen / positive regulation of angiogenesis / antimicrobial humoral immune response mediated by antimicrobial peptide / tertiary granule lumen / cellular response to tumor necrosis factor / antibacterial humoral response / cellular response to lipopolysaccharide / defense response to Gram-negative bacterium / amyloid fibril formation / defense response to Gram-positive bacterium / defense response to bacterium / positive regulation of protein phosphorylation / lipopolysaccharide binding / specific granule lumen / positive regulation of angiogenesis / antimicrobial humoral immune response mediated by antimicrobial peptide / tertiary granule lumen / cellular response to tumor necrosis factor / antibacterial humoral response / cellular response to lipopolysaccharide / defense response to Gram-negative bacterium / amyloid fibril formation / defense response to Gram-positive bacterium / defense response to bacterium / positive regulation of protein phosphorylation /  自然免疫系 / positive regulation of cell population proliferation / Neutrophil degranulation / 自然免疫系 / positive regulation of cell population proliferation / Neutrophil degranulation /  extracellular space / extracellular exosome / extracellular region extracellular space / extracellular exosome / extracellular region類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  溶液NMR / distance geometry 溶液NMR / distance geometry | ||||||
 データ登録者 データ登録者 | Datta, A. / Bhunia, A. | ||||||
 引用 引用 |  ジャーナル: ACS Appl Mater Interfaces / 年: 2017 ジャーナル: ACS Appl Mater Interfaces / 年: 2017タイトル: Conformational Aspects of High Content Packing of Antimicrobial Peptides in Polymer Microgels 著者: Singh, S. / Datta, A. / Borro, B.C. / Davoudi, M. / Schmidtchen, A. / Bhunia, A. / Malmsten, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5xng.cif.gz 5xng.cif.gz | 135.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5xng.ent.gz pdb5xng.ent.gz | 101.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5xng.json.gz 5xng.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xn/5xng https://data.pdbj.org/pub/pdb/validation_reports/xn/5xng ftp://data.pdbj.org/pub/pdb/validation_reports/xn/5xng ftp://data.pdbj.org/pub/pdb/validation_reports/xn/5xng | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  5xrxC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 2179.630 Da / 分子数: 1 / 断片: UNP residues 149-165 / 由来タイプ: 合成 詳細: EFK17 is a truncated part of the human antimicrobial peptide LL37 由来: (合成)   Homo sapiens (ヒト) / 参照: UniProt: P49913 Homo sapiens (ヒト) / 参照: UniProt: P49913 |
|---|
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | タイプ: solution 内容: 1 mM EFK17A, 55.5 mM Water, 50 mM Deuterium oxide, 1 mM TSP, 16 uM Microgel MAA60, 90% H2O/10% D2O Label: EFK17A_1H / 溶媒系: 90% H2O/10% D2O | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||
| 試料状態 | イオン強度: 0 mM / Label: condition_1 / pH: 4.5 / PH err: 0.05 / 圧: 1 atm / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker Ascend TM / 製造業者: Bruker / モデル : Ascend TM / 磁場強度: 800 MHz : Ascend TM / 磁場強度: 800 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: distance geometry / ソフトェア番号: 1 | ||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj


