+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5ol2 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The electron transferring flavoprotein/butyryl-CoA dehydrogenase complex from Clostridium difficile | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード |  FLAVOPROTEIN (フラボタンパク質) / Flavin based electron bifurcation / FLAVOPROTEIN (フラボタンパク質) / Flavin based electron bifurcation /  Bioenergetics / electron transferring flavoprotein / Bioenergetics / electron transferring flavoprotein /  acyl-CoA dehydrogenase (アシルCoAデヒドロゲナーゼ) acyl-CoA dehydrogenase (アシルCoAデヒドロゲナーゼ) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報acryloyl-CoA reductase (NADH) activity /  酸化還元酵素; CH-CH結合に対し酸化酵素として働く; その他の電子受容体を用いる / 酸化還元酵素; CH-CH結合に対し酸化酵素として働く; その他の電子受容体を用いる /  短鎖アシルCoAデヒドロゲナーゼ / 短鎖アシルCoAデヒドロゲナーゼ /  butyryl-CoA dehydrogenase activity / butyryl-CoA dehydrogenase activity /  acyl-CoA dehydrogenase activity / acyl-CoA dehydrogenase activity /  flavin adenine dinucleotide binding / flavin adenine dinucleotide binding /  electron transfer activity electron transfer activity類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Clostridioides difficile (クロストリディオイデス・ディフィシル) Clostridioides difficile (クロストリディオイデス・ディフィシル) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.1 Å 分子置換 / 解像度: 3.1 Å | ||||||||||||
 データ登録者 データ登録者 | Demmer, J.K. / Chowdhury, N.P. / Selmer, T. / Ermler, U. / Buckel, W. | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: The semiquinone swing in the bifurcating electron transferring flavoprotein/butyryl-CoA dehydrogenase complex from Clostridium difficile. 著者: Demmer, J.K. / Pal Chowdhury, N. / Selmer, T. / Ermler, U. / Buckel, W. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5ol2.cif.gz 5ol2.cif.gz | 780.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5ol2.ent.gz pdb5ol2.ent.gz | 649.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5ol2.json.gz 5ol2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ol/5ol2 https://data.pdbj.org/pub/pdb/validation_reports/ol/5ol2 ftp://data.pdbj.org/pub/pdb/validation_reports/ol/5ol2 ftp://data.pdbj.org/pub/pdb/validation_reports/ol/5ol2 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Beg auth comp-ID: MET / Beg label comp-ID: MET
NCSアンサンブル:
|
- 要素
要素
-Electron transfer flavoprotein ... , 2種, 4分子 ADBE
| #1: タンパク質 |  電子伝達フラビンタンパク質 / Electron transfer flavoprotein subunit alpha 電子伝達フラビンタンパク質 / Electron transfer flavoprotein subunit alpha分子量: 35587.957 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Clostridioides difficile (クロストリディオイデス・ディフィシル) Clostridioides difficile (クロストリディオイデス・ディフィシル)遺伝子: etfA, etfA_4, BN1095_140023, BN1096_550022, BN1097_530021, SAMEA3374989_03962, SAMEA3375004_04103, SAMEA3375059_03747 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: A0A031WK27, UniProt: A0A125V455*PLUS Escherichia coli (大腸菌) / 参照: UniProt: A0A031WK27, UniProt: A0A125V455*PLUS#2: タンパク質 |  電子伝達フラビンタンパク質 / Electron transfer flavoprotein subunit beta / Electron transfer flavoproteins subunit beta 電子伝達フラビンタンパク質 / Electron transfer flavoprotein subunit beta / Electron transfer flavoproteins subunit beta分子量: 28346.551 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Clostridioides difficile (クロストリディオイデス・ディフィシル) Clostridioides difficile (クロストリディオイデス・ディフィシル)遺伝子: etfB, etfB_3, etfB_4, BN1095_140022, BN1096_550021, BN1097_530020, IM33_05895, SAMEA3374973_01501, SAMEA3374989_03963, SAMEA3375004_04104, SAMEA3375059_03748 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: A0A031WJM1, UniProt: A0A125V3V6*PLUS Escherichia coli (大腸菌) / 参照: UniProt: A0A031WJM1, UniProt: A0A125V3V6*PLUS |
|---|
-タンパク質 , 1種, 2分子 CF
| #3: タンパク質 |  アシルCoAデヒドロゲナーゼ / Acyl-CoA dehydrogenase / short-chain specific / Butyryl-CoA dehydrogenase アシルCoAデヒドロゲナーゼ / Acyl-CoA dehydrogenase / short-chain specific / Butyryl-CoA dehydrogenase分子量: 41350.234 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Clostridioides difficile (クロストリディオイデス・ディフィシル) Clostridioides difficile (クロストリディオイデス・ディフィシル)遺伝子: acdA, bcd, BN1095_140021, BN1096_550020, BN1097_530019, IM33_05890, SAMEA3374973_01502, SAMEA3374989_03964, SAMEA3375004_04105, SAMEA3375059_03749 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌)参照: UniProt: A0A031WJ47, UniProt: A0A125V4E7*PLUS,  短鎖アシルCoAデヒドロゲナーゼ, 短鎖アシルCoAデヒドロゲナーゼ,  酸化還元酵素; CH-CH結合に対し酸化酵素として働く; その他の電子受容体を用いる 酸化還元酵素; CH-CH結合に対し酸化酵素として働く; その他の電子受容体を用いる |
|---|
-非ポリマー , 3種, 13分子 

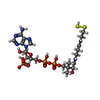


| #4: 化合物 | ChemComp-FAD /  フラビンアデニンジヌクレオチド フラビンアデニンジヌクレオチド#5: 化合物 | ChemComp-CA / #6: 化合物 | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.59 Å3/Da / 溶媒含有率: 73.2 % |
|---|---|
結晶化 | 温度: 277.15 K / 手法: 蒸気拡散法, シッティングドロップ法 詳細: 0.1 M calcium acetate; 14 % (v/v) PEG 400; 0.1 M MES, pH 6.5 |
-データ収集
| 回折 | 平均測定温度: 77 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X10SA / 波長: 1 Å / ビームライン: X10SA / 波長: 1 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2015年11月17日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 3.1→48.866 Å / Num. obs: 70563 / % possible obs: 98.9 % / 冗長度: 7.2 % / Biso Wilson estimate: 104.56 Å2 / Rsym value: 0.143 / Net I/σ(I): 11.1 |
| 反射 シェル | 解像度: 3.1→3.2 Å / 冗長度: 7.6 % / Mean I/σ(I) obs: 0.9 / % possible all: 98.4 |
-位相決定
位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 4KPU and 4L1F 解像度: 3.1→48.866 Å / SU ML: 0.53 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 37.95 / 立体化学のターゲット値: MLHL
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 373.66 Å2 / Biso mean: 130.8201 Å2 / Biso min: 72.55 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 3.1→48.866 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 25
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj



