登録情報 データベース : PDB / ID : 5o46タイトル Crystal structure of Iristatin, a secreted salivary cystatin from the hard tick Ixodes ricinus Iristatin キーワード / / / / 機能・相同性 / / / / / / / / 生物種 Ixodes ricinus (ダニ)手法 / / / 解像度 : 1.76 Å データ登録者 Busa, M. / Rezacova, P. / Kotal, J. / Kotsyfakis, M. / Mares, M. 資金援助 組織 認可番号 国 Czech Science Foundation 13-11043S Ministry of Education, Youth and Sports of the Czech Republic InterBioMed LO1302 INSTITUTE OF ORGANIC CHEMISTRY AND BIOCHEMISTRY AS CR V.V.I. RVO 61388963 Institute of Parasitology, the Czech Academy of Sciences RVO 60077344 Czech Science Foundation P502/12/2409 Grant Agency of the University of South Bohemia in Ceske Budejovice 038/2016/P European Union Marie Curie Reintegration grant PIRG07-GA-2010-268177
ジャーナル : Cell.Mol.Life Sci. / 年 : 2019タイトル : The structure and function of Iristatin, a novel immunosuppressive tick salivary cystatin.著者 : Kotal, J. / Stergiou, N. / Busa, M. / Chlastakova, A. / Berankova, Z. / Rezacova, P. / Langhansova, H. / Schwarz, A. / Calvo, E. / Kopecky, J. / Mares, M. / Schmitt, E. / Chmelar, J. / Kotsyfakis, M. 履歴 登録 2017年5月26日 登録サイト / 処理サイト 改定 1.0 2018年6月13日 Provider / タイプ 改定 1.1 2019年6月26日 Group / Database referencesカテゴリ / citation_author / pdbx_database_procItem _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year 改定 1.2 2024年1月17日 Group / Database references / Refinement descriptionカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード cystatin /
cystatin /  protease inhibitor (プロテアーゼ阻害剤) /
protease inhibitor (プロテアーゼ阻害剤) /  tick (マダニ) /
tick (マダニ) /  saliva (唾液)
saliva (唾液) Cystatin domain / Cystatin-like domain /
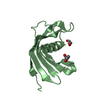
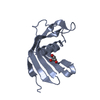
Cystatin domain / Cystatin-like domain /  Cystatin domain / Nuclear Transport Factor 2; Chain: A, - #10 / cysteine-type endopeptidase inhibitor activity / Nuclear Transport Factor 2; Chain: A, / Roll / Alpha Beta / Iristatin
Cystatin domain / Nuclear Transport Factor 2; Chain: A, - #10 / cysteine-type endopeptidase inhibitor activity / Nuclear Transport Factor 2; Chain: A, / Roll / Alpha Beta / Iristatin 機能・相同性情報
機能・相同性情報
 Ixodes ricinus (ダニ)
Ixodes ricinus (ダニ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.76 Å
分子置換 / 解像度: 1.76 Å  データ登録者
データ登録者 チェコ, 7件
チェコ, 7件  引用
引用 ジャーナル: Cell.Mol.Life Sci. / 年: 2019
ジャーナル: Cell.Mol.Life Sci. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5o46.cif.gz
5o46.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5o46.ent.gz
pdb5o46.ent.gz PDB形式
PDB形式 5o46.json.gz
5o46.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/o4/5o46
https://data.pdbj.org/pub/pdb/validation_reports/o4/5o46 ftp://data.pdbj.org/pub/pdb/validation_reports/o4/5o46
ftp://data.pdbj.org/pub/pdb/validation_reports/o4/5o46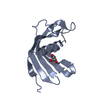
 リンク
リンク 集合体
集合体


 要素
要素
 Ixodes ricinus (ダニ) / 発現宿主:
Ixodes ricinus (ダニ) / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: A0A3B6UEB6*PLUS
Escherichia coli (大腸菌) / 参照: UniProt: A0A3B6UEB6*PLUS グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  BESSY
BESSY  / ビームライン: 14.1 / 波長: 0.9184 Å
/ ビームライン: 14.1 / 波長: 0.9184 Å : 0.9184 Å / 相対比: 1
: 0.9184 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj


