登録情報 データベース : PDB / ID : 5nvaタイトル Substrate-bound outward-open state of a Na+-coupled sialic acid symporter reveals a novel Na+-site Putative sodium:solute symporter キーワード / / / / 機能・相同性 / / / / / / / / 生物種 Proteus mirabilis (バクテリア)手法 / / / 解像度 : 2.26 Å データ登録者 Wahlgren, W.Y. / North, R.A. / Dunevall, E. / Goyal, P. / Grabe, M. / Dobson, R. / Abramson, J. / Ramaswamy, S. / Friemann, R. 資金援助 組織 認可番号 国 Swedish Research Council 2011-5790 Swedish Research Council 2010-1759 Swedish Research Council 221-2013-730 The Swedish Governmental Agency for Innovation Systems (VINNOVA) 2013-04655 Italian Ministry of Instruction University and Research PON01_00937 National Institutes of Health R01GM078844 and R01GM089740 Indo-Swedish grant by the Department of Biotechnology BT/IN/Sweden/41/SR/2013 Royal Society of New Zealand UOC1506 Wellcome Trust WT/099165/Z/12/Z
ジャーナル : Nat Commun / 年 : 2018タイトル : Substrate-bound outward-open structure of a Na+-coupled sialic acid symporter reveals a new Na+site.著者: Wahlgren, W.Y. / Dunevall, E. / North, R.A. / Paz, A. / Scalise, M. / Bisignano, P. / Bengtsson-Palme, J. / Goyal, P. / Claesson, E. / Caing-Carlsson, R. / Andersson, R. / Beis, K. / Nilsson, ... 著者 : Wahlgren, W.Y. / Dunevall, E. / North, R.A. / Paz, A. / Scalise, M. / Bisignano, P. / Bengtsson-Palme, J. / Goyal, P. / Claesson, E. / Caing-Carlsson, R. / Andersson, R. / Beis, K. / Nilsson, U.J. / Farewell, A. / Pochini, L. / Indiveri, C. / Grabe, M. / Dobson, R.C.J. / Abramson, J. / Ramaswamy, S. / Friemann, R. 履歴 登録 2017年5月3日 登録サイト / 処理サイト 改定 1.0 2018年4月4日 Provider / タイプ 改定 1.1 2018年5月16日 Group / Database references / カテゴリ / citation_authorItem _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year 改定 1.2 2019年10月16日 Group / カテゴリ / reflns_shell / Item 改定 1.3 2020年7月29日 Group / Derived calculations / Structure summaryカテゴリ chem_comp / entity ... chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_conn / struct_site / struct_site_gen Item _chem_comp.name / _chem_comp.type ... _chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id 解説 / Provider / タイプ 改定 1.4 2024年5月8日 Group / Database references / Structure summaryカテゴリ chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 Item / _database_2.pdbx_DOI / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード MEMBRANE PROTEIN (膜タンパク質) /
MEMBRANE PROTEIN (膜タンパク質) /  membrane transporter (膜輸送体) /
membrane transporter (膜輸送体) /  sialic acid (シアル酸) / Outward-open / sodium-coupled
sialic acid (シアル酸) / Outward-open / sodium-coupled symporter activity / sodium ion transport /
symporter activity / sodium ion transport /  生体膜 / N-acetyl-beta-neuraminic acid / Putative sodium:solute symporter
生体膜 / N-acetyl-beta-neuraminic acid / Putative sodium:solute symporter 機能・相同性情報
機能・相同性情報
 Proteus mirabilis (バクテリア)
Proteus mirabilis (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.26 Å
分子置換 / 解像度: 2.26 Å  データ登録者
データ登録者 スウェーデン,
スウェーデン,  イタリア,
イタリア,  米国,
米国,  インド,
インド,  ニュージーランド,
ニュージーランド,  英国, 9件
英国, 9件  引用
引用 ジャーナル: Nat Commun / 年: 2018
ジャーナル: Nat Commun / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5nva.cif.gz
5nva.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5nva.ent.gz
pdb5nva.ent.gz PDB形式
PDB形式 5nva.json.gz
5nva.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/nv/5nva
https://data.pdbj.org/pub/pdb/validation_reports/nv/5nva ftp://data.pdbj.org/pub/pdb/validation_reports/nv/5nva
ftp://data.pdbj.org/pub/pdb/validation_reports/nv/5nva リンク
リンク 集合体
集合体
 要素
要素
 Proteus mirabilis (strain HI4320) (バクテリア)
Proteus mirabilis (strain HI4320) (バクテリア)
 Escherichia coli (大腸菌) / 参照: UniProt: B4EZY7
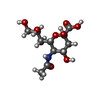
Escherichia coli (大腸菌) / 参照: UniProt: B4EZY7 N-アセチルノイラミン酸
N-アセチルノイラミン酸 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.2 / 波長: 1 Å
/ ビームライン: 5.0.2 / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換 / 解像度: 2.26→82.176 Å / SU ML: 0.32 / 交差検証法: THROUGHOUT / σ(F): 1.33 / 位相誤差: 27.09
分子置換 / 解像度: 2.26→82.176 Å / SU ML: 0.32 / 交差検証法: THROUGHOUT / σ(F): 1.33 / 位相誤差: 27.09  ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj