登録情報 データベース : PDB / ID : 5fv6タイトル KpFlo11 presents a novel member of the Flo11 family with a unique recognition pattern for homophilic interactions Flocculation protein FLO11 キーワード / / / / / / 機能・相同性 / / / / / / / 生物種 Komagataella phaffii (菌類)手法 / / / 解像度 : 2 Å データ登録者 Kraushaar, T. / Brueckner, S. / Mikolaiski, M. / Schreiner, F. / Veelders, M. / Moesch, H.U. / Essen, L.O. ジャーナル : Elife / 年 : 2020タイトル : Kin discrimination in social yeast is mediated by cell surface receptors of the Flo11 adhesin family.著者 : Bruckner, S. / Schubert, R. / Kraushaar, T. / Hartmann, R. / Hoffmann, D. / Jelli, E. / Drescher, K. / Muller, D.J. / Oliver Essen, L. / Mosch, H.U. 履歴 登録 2016年2月3日 登録サイト / 処理サイト 改定 1.0 2017年2月22日 Provider / タイプ 改定 1.1 2021年1月20日 Group / Derived calculations / Otherカテゴリ citation / citation_author ... citation / citation_author / pdbx_database_status / pdbx_struct_conn_angle / struct_conn / struct_site Item _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _pdbx_database_status.status_code_sf / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _struct_conn.pdbx_dist_value / _struct_conn.ptnr2_auth_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id 改定 2.0 2021年6月30日 Group Advisory / Atomic model ... Advisory / Atomic model / Data collection / Database references / Derived calculations / Polymer sequence / Source and taxonomy / Structure summary カテゴリ atom_site / entity ... atom_site / entity / entity_name_com / entity_poly / entity_poly_seq / entity_src_gen / entity_src_nat / pdbx_distant_solvent_atoms / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_poly_seq_scheme / pdbx_struct_assembly / pdbx_struct_assembly_gen / pdbx_struct_assembly_prop / pdbx_struct_conn_angle / pdbx_struct_sheet_hbond / pdbx_unobs_or_zero_occ_residues / pdbx_validate_close_contact / pdbx_validate_torsion / struct_asym / struct_conf / struct_conn / struct_ref / struct_ref_seq / struct_ref_seq_dif / struct_sheet_range / struct_site / struct_site_gen Item _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ... _atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_alt_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.label_seq_id / _atom_site.occupancy / _atom_site.type_symbol / _entity_src_gen.gene_src_common_name / _entity_src_gen.gene_src_strain / _entity_src_gen.pdbx_beg_seq_num / _entity_src_gen.pdbx_end_seq_num / _entity_src_gen.pdbx_gene_src_gene / _entity_src_gen.pdbx_gene_src_scientific_name / _entity_src_gen.pdbx_host_org_scientific_name / _entity_src_gen.pdbx_seq_type / _entity_src_gen.plasmid_name / _pdbx_entity_nonpoly.entity_id / _pdbx_nonpoly_scheme.asym_id / _pdbx_nonpoly_scheme.auth_seq_num / _pdbx_nonpoly_scheme.entity_id / _pdbx_nonpoly_scheme.ndb_seq_num / _pdbx_nonpoly_scheme.pdb_seq_num / _pdbx_nonpoly_scheme.pdb_strand_id / _pdbx_struct_assembly.details / _pdbx_struct_assembly.oligomeric_count / _pdbx_struct_assembly.oligomeric_details / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.value / _pdbx_struct_sheet_hbond.range_1_auth_asym_id / _pdbx_struct_sheet_hbond.range_2_auth_asym_id / _pdbx_validate_close_contact.auth_asym_id_1 / _pdbx_validate_close_contact.auth_asym_id_2 / _pdbx_validate_close_contact.auth_seq_id_2 / _pdbx_validate_torsion.auth_asym_id / _struct_conf.beg_auth_asym_id / _struct_conf.end_auth_asym_id / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_ref_seq_dif.details / _struct_ref_seq_dif.pdbx_pdb_strand_id / _struct_sheet_range.beg_auth_asym_id / _struct_sheet_range.end_auth_asym_id / _struct_site.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_seq_id / _struct_site_gen.auth_asym_id / _struct_site_gen.auth_seq_id / _struct_site_gen.label_asym_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素
 キーワード
キーワード CELL ADHESION (細胞接着) / HOMOPHILIC CELL-CELL ADHESION /
CELL ADHESION (細胞接着) / HOMOPHILIC CELL-CELL ADHESION /  FIBRONECTIN TYPE III DOMAIN (フィブロネクチンIII型ドメイン) /
FIBRONECTIN TYPE III DOMAIN (フィブロネクチンIII型ドメイン) /  FUNGAL CELL WALL (細胞壁) / AROMATIC SURFACE PATCHES / SURFACE-PLASMON RESONANCE SPECTROSCOPY / CELL ADHESIO
FUNGAL CELL WALL (細胞壁) / AROMATIC SURFACE PATCHES / SURFACE-PLASMON RESONANCE SPECTROSCOPY / CELL ADHESIO 酢酸塩 /
酢酸塩 /  Flocculation protein FLO11
Flocculation protein FLO11 機能・相同性情報
機能・相同性情報 Komagataella phaffii (菌類)
Komagataella phaffii (菌類) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Elife / 年: 2020
ジャーナル: Elife / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5fv6.cif.gz
5fv6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5fv6.ent.gz
pdb5fv6.ent.gz PDB形式
PDB形式 5fv6.json.gz
5fv6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/fv/5fv6
https://data.pdbj.org/pub/pdb/validation_reports/fv/5fv6 ftp://data.pdbj.org/pub/pdb/validation_reports/fv/5fv6
ftp://data.pdbj.org/pub/pdb/validation_reports/fv/5fv6 リンク
リンク 集合体
集合体

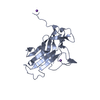
 :
:  要素
要素 / Flo11p / Flocculin-11 / KpFLO11
/ Flo11p / Flocculin-11 / KpFLO11 Komagataella phaffii (strain GS115 / ATCC 20864) (菌類)
Komagataella phaffii (strain GS115 / ATCC 20864) (菌類)
 Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): T7 SHUFFLE EXPRESS / 参照: UniProt: C4R2D7
Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): T7 SHUFFLE EXPRESS / 参照: UniProt: C4R2D7 酢酸塩
酢酸塩 グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 10
X線回折 / 使用した結晶の数: 10  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  BESSY
BESSY  / ビームライン: 14.1 / 波長: 0.97779
/ ビームライン: 14.1 / 波長: 0.97779  : 0.97779 Å / 相対比: 1
: 0.97779 Å / 相対比: 1  解析
解析 :
:  分子置換 / 解像度: 2→40 Å / Cor.coef. Fo:Fc: 0.931 / Cor.coef. Fo:Fc free: 0.898 / SU B: 6.681 / SU ML: 0.178 / 交差検証法: THROUGHOUT / ESU R: 0.247 / ESU R Free: 0.201 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
分子置換 / 解像度: 2→40 Å / Cor.coef. Fo:Fc: 0.931 / Cor.coef. Fo:Fc free: 0.898 / SU B: 6.681 / SU ML: 0.178 / 交差検証法: THROUGHOUT / ESU R: 0.247 / ESU R Free: 0.201 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





