| 非結晶学的対称性 (NCS) | NCS oper: | ID | Code | Matrix | ベクター |
|---|
| 1 | given(1), (1), (1) | | 2 | generate(-0.999996, -0.002302, 0.001795), (-0.002276, 0.999895, 0.014288), (-0.001827, 0.014284, -0.999896)0.51308, 0.23527, -44.55749| 3 | generate(-0.999804, 0.014332, 0.013667), (0.017286, 0.294889, 0.955375), (0.009662, 0.955424, -0.295078)0.6795, 21.00632, -28.46054| 4 | generate(0.999793, -0.016922, -0.011294), (0.016017, 0.31243, 0.949806), (-0.012544, -0.94979, 0.312636)-0.17733, 20.82702, -15.87189| 5 | generate(0.999847, 0.013551, -0.01108), (-0.0147, 0.306336, -0.95181), (-0.009503, 0.951827, 0.306488)-0.20949, -21.4437, -15.12339| 6 | generate(-0.999776, -0.016671, 0.013057), (-0.017743, 0.322893, -0.946269), (0.011559, -0.946289, -0.323117)0.70902, -21.41007, -29.92929| 7 | generate(-0.999555, 0.006623, 0.029099), (0.011319, -0.81808, 0.574992), (0.027614, 0.575066, 0.817641)1.05069, 12.04954, -3.88384| 8 | generate(0.999605, -0.00986, -0.026302), (0.007539, -0.807835, 0.58936), (-0.027059, -0.589326, -0.807442)-0.44777, 12.29299, -40.58977| 9 | generate(-0.999603, -0.009822, 0.026425), (-0.007981, -0.800403, -0.59941), (0.027039, -0.599382, 0.800006)0.96559, -13.96919, -4.84497| 10 | generate(0.999583, 0.009388, -0.027298), (-0.008465, -0.808733, -0.588116), (-0.027598, 0.588102, -0.808316)| -0.65129, -13.8441, -40.2117 | | | | | | | | | | | | | | | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード VIRUS (ウイルス) /
VIRUS (ウイルス) /  Pentamer /
Pentamer /  capsid (カプシド) / honeybee virus /
capsid (カプシド) / honeybee virus /  pathogen (病原体)
pathogen (病原体) 機能・相同性情報
機能・相同性情報
 Israeli acute paralysis virus (腐蛆病)
Israeli acute paralysis virus (腐蛆病) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.7 Å
分子置換 / 解像度: 2.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Virol. / 年: 2016
ジャーナル: J.Virol. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5cdd.cif.gz
5cdd.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5cdd.ent.gz
pdb5cdd.ent.gz PDB形式
PDB形式 5cdd.json.gz
5cdd.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/cd/5cdd
https://data.pdbj.org/pub/pdb/validation_reports/cd/5cdd ftp://data.pdbj.org/pub/pdb/validation_reports/cd/5cdd
ftp://data.pdbj.org/pub/pdb/validation_reports/cd/5cdd リンク
リンク 集合体
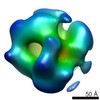
集合体


 : D5 (2回x5回 2面回転対称
: D5 (2回x5回 2面回転対称 ))
)) 要素
要素
 Israeli acute paralysis virus (腐蛆病) / 参照: UniProt: B3TZF1
Israeli acute paralysis virus (腐蛆病) / 参照: UniProt: B3TZF1
 Israeli acute paralysis virus (腐蛆病) / 参照: UniProt: D1FK67
Israeli acute paralysis virus (腐蛆病) / 参照: UniProt: D1FK67
 Israeli acute paralysis virus (腐蛆病) / 参照: UniProt: D1FK67
Israeli acute paralysis virus (腐蛆病) / 参照: UniProt: D1FK67 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06SA / 波長: 0.99999 Å
/ ビームライン: X06SA / 波長: 0.99999 Å : 0.99999 Å / 相対比: 1
: 0.99999 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換 / 解像度: 2.7→70 Å / 交差検証法: FREE R-VALUE / σ(F): 0
分子置換 / 解像度: 2.7→70 Å / 交差検証法: FREE R-VALUE / σ(F): 0  ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj

