| 登録情報 | データベース: PDB / ID: 4mhw
|
|---|
| タイトル | Crystal structure of ThiT with small molecule BAT-25 |
|---|
 要素 要素 | Thiamine transporter ThiT |
|---|
 キーワード キーワード | THIAMINE BINDING PROTEIN / S-component / ECF transporter /  ABC transporter / substrate binding domain for thiamin / ABC transporter / substrate binding domain for thiamin /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) |
|---|
| 機能・相同性 | Arp2/3 complex 21 kDa subunit ARPC3 - #20 / Arp2/3 complex 21 kDa subunit ARPC3 / Orthogonal Bundle / Mainly Alpha / Chem-0YN / DI(HYDROXYETHYL)ETHER / :  機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Lactococcus lactis subsp. cremoris (乳酸菌) Lactococcus lactis subsp. cremoris (乳酸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å |
|---|
 データ登録者 データ登録者 | Swier, L.J.Y.M. / Guskov, A. / Slotboom, D.J. |
|---|
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Published
タイトル: Crystal structure of ThiT with small molecules modulators
著者: Swier, L.J.Y.M. / Gomez, L. / Guskov, A. / Hirsch, A.K.H. / Slotboom, D.J. |
|---|
| 履歴 | | 登録 | 2013年8月30日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年9月3日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年7月29日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp / entity ...chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_ref_seq_dif / struct_site / struct_site_gen
Item: _chem_comp.mon_nstd_flag / _chem_comp.name ..._chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_ref_seq_dif.details
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 1.2 | 2023年9月20日 | Group: Data collection / Database references ...Data collection / Database references / Refinement description / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード ABC transporter / substrate binding domain for thiamin /
ABC transporter / substrate binding domain for thiamin /  TRANSPORT PROTEIN (運搬体タンパク質)
TRANSPORT PROTEIN (運搬体タンパク質) 機能・相同性情報
機能・相同性情報
 Lactococcus lactis subsp. cremoris (乳酸菌)
Lactococcus lactis subsp. cremoris (乳酸菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.5 Å
分子置換 / 解像度: 2.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4mhw.cif.gz
4mhw.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4mhw.ent.gz
pdb4mhw.ent.gz PDB形式
PDB形式 4mhw.json.gz
4mhw.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/mh/4mhw
https://data.pdbj.org/pub/pdb/validation_reports/mh/4mhw ftp://data.pdbj.org/pub/pdb/validation_reports/mh/4mhw
ftp://data.pdbj.org/pub/pdb/validation_reports/mh/4mhw
 リンク
リンク 集合体
集合体
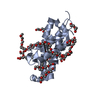

 要素
要素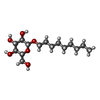


 Lactococcus lactis subsp. cremoris (乳酸菌)
Lactococcus lactis subsp. cremoris (乳酸菌)
 Lactococcus lactis (乳酸菌) / 株 (発現宿主): NZ9000 / 参照: UniProt: D8KFM5
Lactococcus lactis (乳酸菌) / 株 (発現宿主): NZ9000 / 参照: UniProt: D8KFM5













 ポリエチレングリコール
ポリエチレングリコール ポリエチレングリコール
ポリエチレングリコール ジエチレングリコール
ジエチレングリコール ポリエチレングリコール
ポリエチレングリコール ジエチレングリコールモノメチルエーテル
ジエチレングリコールモノメチルエーテル ポリエチレングリコール
ポリエチレングリコール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID29 / 波長: 0.97625 Å
/ ビームライン: ID29 / 波長: 0.97625 Å : 0.97625 Å / 相対比: 1
: 0.97625 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







