+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4m4x | ||||||
|---|---|---|---|---|---|---|---|
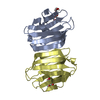
| タイトル | Structure and Dimerization Properties of the Aryl Hydrocarbon Receptor (AHR) PAS-A Domain | ||||||
 要素 要素 | Aryl hydrocarbon receptor 芳香族炭化水素受容体 芳香族炭化水素受容体 | ||||||
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / AHR / PAS-A / Dimer / interface / TRANSCRIPTION (転写 (生物学)) / AHR / PAS-A / Dimer / interface /  transcription factor (転写因子) / ARNT transcription factor (転写因子) / ARNT | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報circumferential growth involved in left ventricle morphogenesis / negative regulation of calcium ion transmembrane transport / cellular response to 3-methylcholanthrene / regulation of heart growth / negative regulation of T cell mediated immune response to tumor cell / cytosolic aryl hydrocarbon receptor complex / glomerulus morphogenesis / gland development / reactive oxygen species biosynthetic process / Phase I - Functionalization of compounds ...circumferential growth involved in left ventricle morphogenesis / negative regulation of calcium ion transmembrane transport / cellular response to 3-methylcholanthrene / regulation of heart growth / negative regulation of T cell mediated immune response to tumor cell / cytosolic aryl hydrocarbon receptor complex / glomerulus morphogenesis / gland development / reactive oxygen species biosynthetic process / Phase I - Functionalization of compounds /  生体異物 / Aryl hydrocarbon receptor signalling / kidney morphogenesis / omega-hydroxylase P450 pathway / arachidonic acid omega-hydroxylase activity / positive regulation of growth rate / lymphocyte homeostasis / 生体異物 / Aryl hydrocarbon receptor signalling / kidney morphogenesis / omega-hydroxylase P450 pathway / arachidonic acid omega-hydroxylase activity / positive regulation of growth rate / lymphocyte homeostasis /  regulation of adaptive immune response / cellular response to 2,3,7,8-tetrachlorodibenzodioxine / Endogenous sterols / nuclear aryl hydrocarbon receptor complex / cardiac left ventricle morphogenesis / negative regulation of osteoblast proliferation / cellular response to toxic substance / reproductive structure development / prostate gland development / regulation of adaptive immune response / cellular response to 2,3,7,8-tetrachlorodibenzodioxine / Endogenous sterols / nuclear aryl hydrocarbon receptor complex / cardiac left ventricle morphogenesis / negative regulation of osteoblast proliferation / cellular response to toxic substance / reproductive structure development / prostate gland development /  aryl hydrocarbon receptor complex / B-1 B cell homeostasis / post-embryonic hemopoiesis / negative regulation of DNA biosynthetic process / camera-type eye development / vasculature development / negative regulation of systemic arterial blood pressure / aryl hydrocarbon receptor complex / B-1 B cell homeostasis / post-embryonic hemopoiesis / negative regulation of DNA biosynthetic process / camera-type eye development / vasculature development / negative regulation of systemic arterial blood pressure /  循環器 / blood vessel morphogenesis / negative regulation of vasoconstriction / branching involved in blood vessel morphogenesis / 循環器 / blood vessel morphogenesis / negative regulation of vasoconstriction / branching involved in blood vessel morphogenesis /  immune system process / blood vessel development / immune system process / blood vessel development /  E-box binding / T cell homeostasis / E-box binding / T cell homeostasis /  aryl hydrocarbon receptor binding / B cell homeostasis / protein localization to nucleus / positive regulation of cell size / aryl hydrocarbon receptor binding / B cell homeostasis / protein localization to nucleus / positive regulation of cell size /  toxic substance binding / negative regulation of osteoblast differentiation / blood vessel remodeling / positive regulation of RNA polymerase II transcription preinitiation complex assembly / cis-regulatory region sequence-specific DNA binding / ovarian follicle development / spleen development / xenobiotic metabolic process / B cell differentiation / liver development / toxic substance binding / negative regulation of osteoblast differentiation / blood vessel remodeling / positive regulation of RNA polymerase II transcription preinitiation complex assembly / cis-regulatory region sequence-specific DNA binding / ovarian follicle development / spleen development / xenobiotic metabolic process / B cell differentiation / liver development /  Hsp90 protein binding / circadian regulation of gene expression / cell morphogenesis / response to organic cyclic compound / Hsp90 protein binding / circadian regulation of gene expression / cell morphogenesis / response to organic cyclic compound /  nuclear receptor activity / sequence-specific double-stranded DNA binding / protein-folding chaperone binding / nuclear receptor activity / sequence-specific double-stranded DNA binding / protein-folding chaperone binding /  transcription regulator complex / sequence-specific DNA binding / transcription by RNA polymerase II / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / response to xenobiotic stimulus / positive regulation of apoptotic process / transcription regulator complex / sequence-specific DNA binding / transcription by RNA polymerase II / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / response to xenobiotic stimulus / positive regulation of apoptotic process /  細胞周期 / protein heterodimerization activity / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / 細胞周期 / protein heterodimerization activity / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / protein homodimerization activity /  DNA binding / identical protein binding / DNA binding / identical protein binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.551 Å 分子置換 / 解像度: 2.551 Å | ||||||
 データ登録者 データ登録者 | Wu, D. / Potluri, N. / Kim, Y. / Rastinejad, F. | ||||||
 引用 引用 |  ジャーナル: Mol.Cell.Biol. / 年: 2013 ジャーナル: Mol.Cell.Biol. / 年: 2013タイトル: Structure and dimerization properties of the aryl hydrocarbon receptor PAS-A domain. 著者: Wu, D. / Potluri, N. / Kim, Y. / Rastinejad, F. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4m4x.cif.gz 4m4x.cif.gz | 111.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4m4x.ent.gz pdb4m4x.ent.gz | 85.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4m4x.json.gz 4m4x.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/m4/4m4x https://data.pdbj.org/pub/pdb/validation_reports/m4/4m4x ftp://data.pdbj.org/pub/pdb/validation_reports/m4/4m4x ftp://data.pdbj.org/pub/pdb/validation_reports/m4/4m4x | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン: (詳細: chain B and segid A) NCSドメイン領域: (Selection details: chain 'B' and segid 'A ') |
- 要素
要素
| #1: タンパク質 |  芳香族炭化水素受容体 / Ah receptor / AhR 芳香族炭化水素受容体 / Ah receptor / AhR分子量: 21129.623 Da / 分子数: 2 / 断片: PAS-A domain, UNP residues 110-267 / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) / 株: C57BL / 遺伝子: Ahr / プラスミド: pSJ2 / 発現宿主: Mus musculus (ハツカネズミ) / 株: C57BL / 遺伝子: Ahr / プラスミド: pSJ2 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: P30561 Escherichia coli (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: P30561#2: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.53 Å3/Da / 溶媒含有率: 51.32 % |
|---|---|
結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 100 mM HEPES, 1.6 M Ammonium Sulfate and 2% PEG 1000, pH 7.5, VAPOR DIFFUSION, SITTING DROP, temperature 277K |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 0.9793 Å / ビームライン: 19-ID / 波長: 0.9793 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.9793 Å / 相対比: 1 : 0.9793 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.55→50 Å / Num. obs: 14659 / % possible obs: 99.5 % / 冗長度: 9.1 % / Rmerge(I) obs: 0.07 / Χ2: 1.124 / Net I/σ(I): 11.2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル |
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 2.551→33.86 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.8256 / SU ML: 0.26 / σ(F): 1.35 / 位相誤差: 23.56 / 立体化学のターゲット値: ML 分子置換 / 解像度: 2.551→33.86 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.8256 / SU ML: 0.26 / σ(F): 1.35 / 位相誤差: 23.56 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 160.61 Å2 / Biso mean: 60.8807 Å2 / Biso min: 25.76 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.551→33.86 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | 数: 929 / タイプ: POSITIONAL / Rms dev position: 12.282 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 5
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj








