登録情報 データベース : PDB / ID : 3ud8タイトル Crystal Structure Analysis of FGF1-Disaccharide(NI22) complex Heparin-binding growth factor 1 キーワード / 機能・相同性 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 2.37 Å データ登録者 Hung, S.-C. / Shi, Z. ジャーナル : J.Am.Chem.Soc. / 年 : 2012タイトル : Divergent synthesis of 48 heparan sulfate-based disaccharides and probing the specific sugar-fibroblast growth factor-1 interaction著者 : Hu, Y.-P. / Zhong, Y.-Q. / Chen, Z.-G. / Chen, C.-Y. / Shi, Z. / Zulueta, M.M.L. / Ku, C.-C. / Lee, P.-Y. / Wang, C.-C. / Hung, S.-C. 履歴 登録 2011年10月27日 登録サイト / 処理サイト 改定 1.0 2012年11月28日 Provider / タイプ 改定 1.1 2013年1月16日 Group 改定 2.0 2020年7月29日 Group Advisory / Atomic model ... Advisory / Atomic model / Data collection / Database references / Derived calculations / Structure summary カテゴリ atom_site / atom_site_anisotrop ... atom_site / atom_site_anisotrop / chem_comp / database_PDB_caveat / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_validate_chiral / struct_asym / struct_conn / struct_ref_seq_dif / struct_site / struct_site_gen Item _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ... _atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _atom_site_anisotrop.U[1][1] / _atom_site_anisotrop.U[1][2] / _atom_site_anisotrop.U[1][3] / _atom_site_anisotrop.U[2][2] / _atom_site_anisotrop.U[2][3] / _atom_site_anisotrop.U[3][3] / _atom_site_anisotrop.pdbx_auth_asym_id / _atom_site_anisotrop.pdbx_auth_atom_id / _atom_site_anisotrop.pdbx_auth_comp_id / _atom_site_anisotrop.pdbx_auth_seq_id / _atom_site_anisotrop.pdbx_label_asym_id / _atom_site_anisotrop.pdbx_label_atom_id / _atom_site_anisotrop.pdbx_label_comp_id / _atom_site_anisotrop.type_symbol / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_validate_chiral.auth_asym_id / _pdbx_validate_chiral.auth_seq_id / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_ref_seq_dif.details 解説 / Provider / タイプ 改定 2.1 2023年11月1日 Group Data collection / Database references ... Data collection / Database references / Refinement description / Structure summary カテゴリ chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_DOI / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HORMONE (ホルモン) / Heparin/Heparan sulfate Binding
HORMONE (ホルモン) / Heparin/Heparan sulfate Binding 機能・相同性情報
機能・相同性情報 fibroblast growth factor receptor binding / FGFR2c ligand binding and activation / Activated point mutants of FGFR2 / Phospholipase C-mediated cascade; FGFR2 / FGFR4 ligand binding and activation / FGFR1b ligand binding and activation / Phospholipase C-mediated cascade; FGFR4 / Signaling by activated point mutants of FGFR1 / FGFR1c ligand binding and activation / organ induction / Downstream signaling of activated FGFR1 / Phospholipase C-mediated cascade: FGFR1 / positive regulation of hepatocyte proliferation / S100 protein binding / positive regulation of intracellular signal transduction / Signaling by FGFR2 IIIa TM / PI-3K cascade:FGFR3 / positive regulation of sprouting angiogenesis / PI-3K cascade:FGFR2 / PI-3K cascade:FGFR4 / PI-3K cascade:FGFR1 / positive regulation of cell division / PI3K Cascade / anatomical structure morphogenesis / fibroblast growth factor receptor signaling pathway / SHC-mediated cascade:FGFR3 / SHC-mediated cascade:FGFR2 / SHC-mediated cascade:FGFR4 / SHC-mediated cascade:FGFR1 / FRS-mediated FGFR3 signaling / FRS-mediated FGFR2 signaling / FRS-mediated FGFR4 signaling / Signaling by FGFR3 in disease / FRS-mediated FGFR1 signaling /
fibroblast growth factor receptor binding / FGFR2c ligand binding and activation / Activated point mutants of FGFR2 / Phospholipase C-mediated cascade; FGFR2 / FGFR4 ligand binding and activation / FGFR1b ligand binding and activation / Phospholipase C-mediated cascade; FGFR4 / Signaling by activated point mutants of FGFR1 / FGFR1c ligand binding and activation / organ induction / Downstream signaling of activated FGFR1 / Phospholipase C-mediated cascade: FGFR1 / positive regulation of hepatocyte proliferation / S100 protein binding / positive regulation of intracellular signal transduction / Signaling by FGFR2 IIIa TM / PI-3K cascade:FGFR3 / positive regulation of sprouting angiogenesis / PI-3K cascade:FGFR2 / PI-3K cascade:FGFR4 / PI-3K cascade:FGFR1 / positive regulation of cell division / PI3K Cascade / anatomical structure morphogenesis / fibroblast growth factor receptor signaling pathway / SHC-mediated cascade:FGFR3 / SHC-mediated cascade:FGFR2 / SHC-mediated cascade:FGFR4 / SHC-mediated cascade:FGFR1 / FRS-mediated FGFR3 signaling / FRS-mediated FGFR2 signaling / FRS-mediated FGFR4 signaling / Signaling by FGFR3 in disease / FRS-mediated FGFR1 signaling /  Hsp70 protein binding / Signaling by FGFR2 in disease / Signaling by FGFR1 in disease / activation of protein kinase B activity / positive regulation of endothelial cell migration /
Hsp70 protein binding / Signaling by FGFR2 in disease / Signaling by FGFR1 in disease / activation of protein kinase B activity / positive regulation of endothelial cell migration /  細胞外マトリックス / epithelial cell proliferation / positive regulation of epithelial cell proliferation / Negative regulation of FGFR3 signaling / Negative regulation of FGFR2 signaling / Negative regulation of FGFR4 signaling / Negative regulation of FGFR1 signaling / animal organ morphogenesis /
細胞外マトリックス / epithelial cell proliferation / positive regulation of epithelial cell proliferation / Negative regulation of FGFR3 signaling / Negative regulation of FGFR2 signaling / Negative regulation of FGFR4 signaling / Negative regulation of FGFR1 signaling / animal organ morphogenesis /  growth factor activity / positive regulation of MAP kinase activity / lung development /
growth factor activity / positive regulation of MAP kinase activity / lung development /  wound healing / positive regulation of angiogenesis / Constitutive Signaling by Aberrant PI3K in Cancer /
wound healing / positive regulation of angiogenesis / Constitutive Signaling by Aberrant PI3K in Cancer /  integrin binding / PIP3 activates AKT signaling /
integrin binding / PIP3 activates AKT signaling /  heparin binding / cellular response to heat /
heparin binding / cellular response to heat /  細胞皮質 / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / RAF/MAP kinase cascade /
細胞皮質 / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / RAF/MAP kinase cascade /  血管新生 / positive regulation of ERK1 and ERK2 cascade /
血管新生 / positive regulation of ERK1 and ERK2 cascade /  細胞分化 / positive regulation of cell migration / positive regulation of cell population proliferation / positive regulation of gene expression /
細胞分化 / positive regulation of cell migration / positive regulation of cell population proliferation / positive regulation of gene expression /  シグナル伝達 / positive regulation of transcription by RNA polymerase II /
シグナル伝達 / positive regulation of transcription by RNA polymerase II /  extracellular space / extracellular region /
extracellular space / extracellular region /  核質 /
核質 /  細胞核 /
細胞核 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.37 Å
分子置換 / 解像度: 2.37 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2012
ジャーナル: J.Am.Chem.Soc. / 年: 2012 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3ud8.cif.gz
3ud8.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3ud8.ent.gz
pdb3ud8.ent.gz PDB形式
PDB形式 3ud8.json.gz
3ud8.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ud/3ud8
https://data.pdbj.org/pub/pdb/validation_reports/ud/3ud8 ftp://data.pdbj.org/pub/pdb/validation_reports/ud/3ud8
ftp://data.pdbj.org/pub/pdb/validation_reports/ud/3ud8 リンク
リンク 集合体
集合体
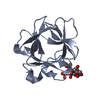


 要素
要素
 Homo sapiens (ヒト) / 遺伝子: FGF1 / プラスミド: pET21b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: FGF1 / プラスミド: pET21b / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P05230
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P05230 オリゴ糖 / 分子量: 609.512 Da / 分子数: 1 / 由来タイプ: 組換発現
オリゴ糖 / 分子量: 609.512 Da / 分子数: 1 / 由来タイプ: 組換発現 リン酸塩
リン酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  NSRRC
NSRRC  / ビームライン: BL13B1 / 波長: 1 Å
/ ビームライン: BL13B1 / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj






