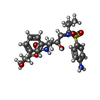
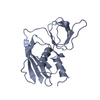
登録情報 データベース : PDB / ID : 3sm2タイトル The crystal structure of XMRV protease complexed with Amprenavir gag-pro-pol polyprotein キーワード / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 手法 / / / 解像度 : 1.75 Å データ登録者 Li, M. / Gustchina, A. / Wlodawer, A. ジャーナル : Febs J. / 年 : 2011タイトル : Structural and biochemical characterization of the inhibitor complexes of xenotropic murine leukemia virus-related virus protease.著者 : Li, M. / Gustchina, A. / Matuz, K. / Tozser, J. / Namwong, S. / Goldfarb, N.E. / Dunn, B.M. / Wlodawer, A. 履歴 登録 2011年6月27日 登録サイト / 処理サイト 改定 1.0 2011年10月12日 Provider / タイプ 改定 1.1 2012年4月4日 Group 改定 1.2 2024年2月28日 Group / Database references / Derived calculationsカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / struct_ref_seq_dif / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id 改定 1.3 2024年3月13日 Group / Structure summary / カテゴリ / pdbx_entity_src_syn / Item
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード Beta-sheet (Βシート) /
Beta-sheet (Βシート) /  Protease (プロテアーゼ) /
Protease (プロテアーゼ) /  Amprenavir /
Amprenavir /  virus (ウイルス) / hydrolase-hydrolase inhibitor complex
virus (ウイルス) / hydrolase-hydrolase inhibitor complex 機能・相同性情報
機能・相同性情報 virion assembly /
virion assembly /  virion component / host multivesicular body / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency /
virion component / host multivesicular body / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency /  RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / DNA recombination ...host cell late endosome membrane /
RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / DNA recombination ...host cell late endosome membrane /  virion assembly /
virion assembly /  virion component / host multivesicular body / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency /
virion component / host multivesicular body / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency /  RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / DNA recombination / structural constituent of virion / aspartic-type endopeptidase activity /
RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / DNA recombination / structural constituent of virion / aspartic-type endopeptidase activity /  DNA-directed DNA polymerase activity / symbiont entry into host cell / host cell plasma membrane /
DNA-directed DNA polymerase activity / symbiont entry into host cell / host cell plasma membrane /  タンパク質分解 /
タンパク質分解 /  DNA binding /
DNA binding /  RNA binding / zinc ion binding /
RNA binding / zinc ion binding /  細胞膜
細胞膜 DG-75 Murine leukemia virus (ウイルス)
DG-75 Murine leukemia virus (ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 1.75 Å
フーリエ合成 / 解像度: 1.75 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Febs J. / 年: 2011
ジャーナル: Febs J. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3sm2.cif.gz
3sm2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3sm2.ent.gz
pdb3sm2.ent.gz PDB形式
PDB形式 3sm2.json.gz
3sm2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/sm/3sm2
https://data.pdbj.org/pub/pdb/validation_reports/sm/3sm2 ftp://data.pdbj.org/pub/pdb/validation_reports/sm/3sm2
ftp://data.pdbj.org/pub/pdb/validation_reports/sm/3sm2 リンク
リンク 集合体
集合体
 要素
要素 DG-75 Murine leukemia virus (ウイルス)
DG-75 Murine leukemia virus (ウイルス)
 Escherichia coli (大腸菌) / 株 (発現宿主): Bl21(de3)tona Prare / 参照: UniProt: Q9E7M1
Escherichia coli (大腸菌) / 株 (発現宿主): Bl21(de3)tona Prare / 参照: UniProt: Q9E7M1 Amprenavir
Amprenavir 水
水 X線回折
X線回折 試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-ID / 波長: 1 Å
/ ビームライン: 22-ID / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  フーリエ合成 / 解像度: 1.75→27.77 Å / Cor.coef. Fo:Fc: 0.966 / Cor.coef. Fo:Fc free: 0.951 / SU B: 2.694 / SU ML: 0.087 / 交差検証法: THROUGHOUT / ESU R Free: 0.128 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
フーリエ合成 / 解像度: 1.75→27.77 Å / Cor.coef. Fo:Fc: 0.966 / Cor.coef. Fo:Fc free: 0.951 / SU B: 2.694 / SU ML: 0.087 / 交差検証法: THROUGHOUT / ESU R Free: 0.128 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj