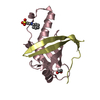
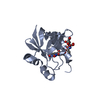
登録情報 データベース : PDB / ID : 3hqcタイトル Crystal structure of Phosphotyrosine-binding domain from the Human Tensin-like C1 domain-containing phosphatase (TENC1) Tensin-like C1 domain-containing phosphatase キーワード / / / / / / / / / / / / / / / / / / / / 機能・相同性 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 1.8 Å データ登録者 Sampathkumar, P. / Romero, R. / Wasserman, S. / Do, J. / Dickey, M. / Bain, K. / Gheyi, T. / Klemke, R. / Atwell, S. / Sauder, J.M. ...Sampathkumar, P. / Romero, R. / Wasserman, S. / Do, J. / Dickey, M. / Bain, K. / Gheyi, T. / Klemke, R. / Atwell, S. / Sauder, J.M. / Burley, S.K. / New York SGX Research Center for Structural Genomics (NYSGXRC) ジャーナル : To be Published タイトル : Crystal structure of Phosphotyrosine-binding domain from the Human Tensin-like C1 domain-containing phosphatase (TENC1)著者 : Sampathkumar, P. / Romero, R. / Wasserman, S. / Do, J. / Dickey, M. / Bain, K. / Gheyi, T. / Klemke, R. / Atwell, S. / Sauder, J.M. / Burley, S.K. 履歴 登録 2009年6月5日 登録サイト / 処理サイト 改定 1.0 2009年7月21日 Provider / タイプ 改定 1.1 2011年7月13日 Group / Version format compliance改定 1.2 2018年11月21日 Group / Structure summary / カテゴリ / Item 改定 1.3 2021年2月10日 Group / Derived calculations / Structure summaryカテゴリ audit_author / citation_author ... audit_author / citation_author / struct_conn / struct_site Item _audit_author.identifier_ORCID / _citation_author.identifier_ORCID ... _audit_author.identifier_ORCID / _citation_author.identifier_ORCID / _struct_conn.pdbx_leaving_atom_flag / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id 改定 1.4 2021年10月13日 Group / カテゴリ / struct_ref_seq_difItem / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details改定 1.5 2023年9月6日 Group / Refinement descriptionカテゴリ / chem_comp_bond / pdbx_initial_refinement_model改定 1.6 2023年11月22日 Group / カテゴリ / chem_comp_bond / Item / _chem_comp_bond.atom_id_2
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HYDROLASE (加水分解酵素) / Human Tensin-like C1 domain-containing phosphatase / TENC1 / Phosphotyrosine Binding Domain / PTB / TNS2 / KIAA1075 /
HYDROLASE (加水分解酵素) / Human Tensin-like C1 domain-containing phosphatase / TENC1 / Phosphotyrosine Binding Domain / PTB / TNS2 / KIAA1075 /  STRUCTURAL GENOMICS (構造ゲノミクス) / PSI-2 /
STRUCTURAL GENOMICS (構造ゲノミクス) / PSI-2 /  PROTEIN STRUCTURE INITIATIVE / NEW YORK SGX Research Center for Structural Genomics / NYSGXRC /
PROTEIN STRUCTURE INITIATIVE / NEW YORK SGX Research Center for Structural Genomics / NYSGXRC /  Cell junction (細胞結合) /
Cell junction (細胞結合) /  Cell membrane (細胞膜) /
Cell membrane (細胞膜) /  Membrane (生体膜) / Metal-binding / Phorbol-ester binding /
Membrane (生体膜) / Metal-binding / Phorbol-ester binding /  Phosphoprotein /
Phosphoprotein /  Protein phosphatase (プロテインホスファターゼ) /
Protein phosphatase (プロテインホスファターゼ) /  SH2 domain (SH2ドメイン) /
SH2 domain (SH2ドメイン) /  Zinc-finger (ジンクフィンガー)
Zinc-finger (ジンクフィンガー) 機能・相同性情報
機能・相同性情報 プロテインチロシンホスファターゼ /
プロテインチロシンホスファターゼ /  protein tyrosine phosphatase activity /
protein tyrosine phosphatase activity /  kidney development / multicellular organism growth ...multicellular organismal-level homeostasis / collagen metabolic process / cellular homeostasis / response to muscle activity / peptidyl-tyrosine dephosphorylation / negative regulation of insulin receptor signaling pathway /
kidney development / multicellular organism growth ...multicellular organismal-level homeostasis / collagen metabolic process / cellular homeostasis / response to muscle activity / peptidyl-tyrosine dephosphorylation / negative regulation of insulin receptor signaling pathway /  プロテインチロシンホスファターゼ /
プロテインチロシンホスファターゼ /  protein tyrosine phosphatase activity /
protein tyrosine phosphatase activity /  kidney development / multicellular organism growth /
kidney development / multicellular organism growth /  kinase binding / negative regulation of cell population proliferation /
kinase binding / negative regulation of cell population proliferation /  focal adhesion /
focal adhesion /  lipid binding / identical protein binding /
lipid binding / identical protein binding /  metal ion binding /
metal ion binding /  細胞膜 /
細胞膜 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.8 Å
分子置換 / 解像度: 1.8 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3hqc.cif.gz
3hqc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3hqc.ent.gz
pdb3hqc.ent.gz PDB形式
PDB形式 3hqc.json.gz
3hqc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/hq/3hqc
https://data.pdbj.org/pub/pdb/validation_reports/hq/3hqc ftp://data.pdbj.org/pub/pdb/validation_reports/hq/3hqc
ftp://data.pdbj.org/pub/pdb/validation_reports/hq/3hqc
 リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: KIAA1075, TENC1, TNS2 / プラスミド: BC-pSGX3(BC); modified pET26b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: KIAA1075, TENC1, TNS2 / プラスミド: BC-pSGX3(BC); modified pET26b / 発現宿主: 
 Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3)-Codon+RIL
Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3)-Codon+RIL 加水分解酵素; エステル加水分解酵素; 1価のリン酸エステル加水分解酵素
加水分解酵素; エステル加水分解酵素; 1価のリン酸エステル加水分解酵素 硫酸塩
硫酸塩 酢酸塩
酢酸塩 グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 31-ID / 波長: 0.9793 Å
/ ビームライン: 31-ID / 波長: 0.9793 Å : 0.9793 Å / 相対比: 1
: 0.9793 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj















