+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3cx9 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal Structure of Human serum albumin complexed with Myristic acid and lysophosphatidylethanolamine | ||||||
 要素 要素 | Serum albumin | ||||||
 キーワード キーワード |  STRUCTURAL PROTEIN (タンパク質) / STRUCTURAL PROTEIN (タンパク質) /  Human serum albumin / Lysophospholipids / Lysophosphatidylethanolamine / Human serum albumin / Lysophospholipids / Lysophosphatidylethanolamine /  Fluorescence quenching (消光) / Cleavage on pair of basic residues / Disease mutation / Fluorescence quenching (消光) / Cleavage on pair of basic residues / Disease mutation /  Glycation (糖化反応) / Glycation (糖化反応) /  Glycoprotein (糖タンパク質) / Lipid-binding / Metal-binding / Glycoprotein (糖タンパク質) / Lipid-binding / Metal-binding /  Secreted (分泌) Secreted (分泌) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to calcium ion starvation /  exogenous protein binding / Ciprofloxacin ADME / exogenous protein binding / Ciprofloxacin ADME /  enterobactin binding / enterobactin binding /  ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME ...cellular response to calcium ion starvation / ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME ...cellular response to calcium ion starvation /  exogenous protein binding / Ciprofloxacin ADME / exogenous protein binding / Ciprofloxacin ADME /  enterobactin binding / enterobactin binding /  ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME / ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME /  antioxidant activity / antioxidant activity /  toxic substance binding / toxic substance binding /  small molecule binding / Scavenging of heme from plasma / Recycling of bile acids and salts / cellular response to starvation / platelet alpha granule lumen / small molecule binding / Scavenging of heme from plasma / Recycling of bile acids and salts / cellular response to starvation / platelet alpha granule lumen /  fatty acid binding / fatty acid binding /  Post-translational protein phosphorylation / Cytoprotection by HMOX1 / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / Post-translational protein phosphorylation / Cytoprotection by HMOX1 / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /  pyridoxal phosphate binding / Platelet degranulation / protein-folding chaperone binding / blood microparticle / copper ion binding / pyridoxal phosphate binding / Platelet degranulation / protein-folding chaperone binding / blood microparticle / copper ion binding /  小胞体 / 小胞体 /  ゴルジ体 / ゴルジ体 /  小胞体 / protein-containing complex / 小胞体 / protein-containing complex /  DNA binding / DNA binding /  extracellular space / extracellular exosome / extracellular region / identical protein binding / extracellular space / extracellular exosome / extracellular region / identical protein binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Guo, S. / Yang, F. / Chen, L. / Bian, C. / Huang, M. | ||||||
 引用 引用 |  ジャーナル: Biochem.J. / 年: 2009 ジャーナル: Biochem.J. / 年: 2009タイトル: Structural basis of transport of lysophospholipids by human serum albumin. 著者: Guo, S. / Shi, X. / Yang, F. / Chen, L. / Meehan, E.J. / Bian, C. / Huang, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3cx9.cif.gz 3cx9.cif.gz | 127.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3cx9.ent.gz pdb3cx9.ent.gz | 97 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3cx9.json.gz 3cx9.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cx/3cx9 https://data.pdbj.org/pub/pdb/validation_reports/cx/3cx9 ftp://data.pdbj.org/pub/pdb/validation_reports/cx/3cx9 ftp://data.pdbj.org/pub/pdb/validation_reports/cx/3cx9 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1n5uS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 |  分子量: 66271.898 Da / 分子数: 1 / 断片: UNP residues 27-608 / 由来タイプ: 天然 / 詳細: human serum albumin / 由来: (天然)   Homo sapiens (ヒト) / 参照: UniProt: P02768 Homo sapiens (ヒト) / 参照: UniProt: P02768 | ||
|---|---|---|---|
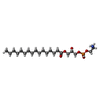
| #2: 化合物 | ChemComp-MYR /  ミリスチン酸 ミリスチン酸#3: 化合物 | ChemComp-LPX / ( | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.5 Å3/Da / 溶媒含有率: 50.87 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 35% PEG 3350, 85mM potassium phosphate, 5mM sodium azide, pH7.5, VAPOR DIFFUSION, SITTING DROP, temperature 298.0K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 22-ID / 波長: 1 Å / ビームライン: 22-ID / 波長: 1 Å |
| 検出器 | タイプ: BRUKER SMART 2000 / 検出器: CCD / 日付: 2007年1月1日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→50 Å / Num. obs: 15244 / % possible obs: 79.1 % / 冗長度: 2 % / Rmerge(I) obs: 0.057 |
| 反射 シェル | 解像度: 2.8→2.873 Å / 冗長度: 1.9 % / Rmerge(I) obs: 0.356 / Num. unique all: 1095 / % possible all: 85.7 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1N5U 解像度: 2.8→50 Å / Cor.coef. Fo:Fc: 0.931 / Cor.coef. Fo:Fc free: 0.866 / SU B: 41.451 / SU ML: 0.366 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R Free: 0.469 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 56.231 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→50 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.873 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj