| 登録情報 | データベース: PDB / ID: 2xk0
|
|---|
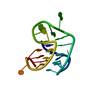
| タイトル | Solution structure of the Tudor domain from Drosophila Polycomblike (Pcl) |
|---|
 要素 要素 | POLYCOMB PROTEIN PCL ポリコーム群タンパク質 ポリコーム群タンパク質 |
|---|
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / AROMATIC CAGE TRANSCRIPTION (転写 (生物学)) / AROMATIC CAGE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
ventral cord development /  多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization / 多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization /  microtubule binding / microtubule binding /  遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription / 遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /  chromatin binding ...ventral cord development / chromatin binding ...ventral cord development /  多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization / 多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization /  microtubule binding / microtubule binding /  遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription / 遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /  chromatin binding / regulation of transcription by RNA polymerase II / chromatin binding / regulation of transcription by RNA polymerase II /  DNA binding / DNA binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 Polycomb-like MTF2 factor 2, C-terminal domain / Polycomb-like MTF2 factor 2 / Set1/Ash2 histone methyltransferase complex subunit ASH2-like, WH /  Tudor domain / Tudor domain /  Tudor domain / SH3 type barrels. - #140 / Tudor domain / SH3 type barrels. - #140 /  Zinc finger, PHD-type, conserved site / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / Zinc finger, PHD-type, conserved site / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. /  Zinc finger, PHD-finger ...Polycomb-like MTF2 factor 2, C-terminal domain / Polycomb-like MTF2 factor 2 / Set1/Ash2 histone methyltransferase complex subunit ASH2-like, WH / Zinc finger, PHD-finger ...Polycomb-like MTF2 factor 2, C-terminal domain / Polycomb-like MTF2 factor 2 / Set1/Ash2 histone methyltransferase complex subunit ASH2-like, WH /  Tudor domain / Tudor domain /  Tudor domain / SH3 type barrels. - #140 / Tudor domain / SH3 type barrels. - #140 /  Zinc finger, PHD-type, conserved site / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / Zinc finger, PHD-type, conserved site / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. /  Zinc finger, PHD-finger / Zinc finger, PHD-finger /  Zinc finger, PHD-type / PHD zinc finger / Zinc finger, PHD-type / PHD zinc finger /  Zinc finger, FYVE/PHD-type / SH3 type barrels. / Zinc finger, FYVE/PHD-type / SH3 type barrels. /  Zinc finger, RING/FYVE/PHD-type / Roll / Mainly Beta類似検索 - ドメイン・相同性 Zinc finger, RING/FYVE/PHD-type / Roll / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |    DROSOPHILA MELANOGASTER (キイロショウジョウバエ) DROSOPHILA MELANOGASTER (キイロショウジョウバエ) |
|---|
| 手法 |  溶液NMR / CYANA 溶液NMR / CYANA |
|---|
 データ登録者 データ登録者 | Friberg, A. / Oddone, A. / Klymenko, T. / Mueller, J. / Sattler, M. |
|---|
 引用 引用 |  ジャーナル: Protein Sci. / 年: 2010 ジャーナル: Protein Sci. / 年: 2010
タイトル: Structure of an Atypical Tudor Domain in the Drosophila Polycomblike Protein
著者: Friberg, A. / Oddone, A. / Klymenko, T. / Mueller, J. / Sattler, M. |
|---|
| 履歴 | | 登録 | 2010年7月7日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2010年8月11日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年5月8日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2020年1月15日 | Group: Data collection / Other / カテゴリ: pdbx_database_status / pdbx_nmr_software
Item: _pdbx_database_status.status_code_cs / _pdbx_database_status.status_code_mr / _pdbx_nmr_software.name |
|---|
| 改定 1.4 | 2021年6月23日 | Group: Data collection / カテゴリ: pdbx_nmr_spectrometer |
|---|
| 改定 1.5 | 2023年6月14日 | Group: Database references / Other / カテゴリ: database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 ポリコーム群タンパク質
ポリコーム群タンパク質  キーワード
キーワード TRANSCRIPTION (転写 (生物学)) / AROMATIC CAGE
TRANSCRIPTION (転写 (生物学)) / AROMATIC CAGE 機能・相同性情報
機能・相同性情報 多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization /
多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization /  microtubule binding /
microtubule binding /  遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /
遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /  chromatin binding ...ventral cord development /
chromatin binding ...ventral cord development /  多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization /
多糸染色体 / anterior/posterior axis specification / defense response to fungus / chromatin organization /  microtubule binding /
microtubule binding /  遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /
遺伝子発現の調節 / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /  chromatin binding / regulation of transcription by RNA polymerase II /
chromatin binding / regulation of transcription by RNA polymerase II /  DNA binding /
DNA binding /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞質
細胞質
 DROSOPHILA MELANOGASTER (キイロショウジョウバエ)
DROSOPHILA MELANOGASTER (キイロショウジョウバエ) 溶液NMR / CYANA
溶液NMR / CYANA  データ登録者
データ登録者 引用
引用 ジャーナル: Protein Sci. / 年: 2010
ジャーナル: Protein Sci. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2xk0.cif.gz
2xk0.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2xk0.ent.gz
pdb2xk0.ent.gz PDB形式
PDB形式 2xk0.json.gz
2xk0.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/xk/2xk0
https://data.pdbj.org/pub/pdb/validation_reports/xk/2xk0 ftp://data.pdbj.org/pub/pdb/validation_reports/xk/2xk0
ftp://data.pdbj.org/pub/pdb/validation_reports/xk/2xk0 リンク
リンク 集合体
集合体
 要素
要素 ポリコーム群タンパク質 / POLYCOMBLIKE PROTEIN
ポリコーム群タンパク質 / POLYCOMBLIKE PROTEIN
 DROSOPHILA MELANOGASTER (キイロショウジョウバエ)
DROSOPHILA MELANOGASTER (キイロショウジョウバエ)
 ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) PLYSS / 参照: UniProt: Q24459
ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) PLYSS / 参照: UniProt: Q24459 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj