+ データを開く
データを開く
- 基本情報
基本情報
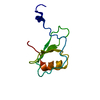
| 登録情報 | データベース: PDB / ID: 2l9r | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Solution NMR Structure of Homeobox domain of Homeobox protein Nkx-3.1 from homo sapiens, Northeast Structural Genomics Consortium Target HR6470A | ||||||
 要素 要素 | Homeobox protein Nkx-3.1 ホメオボックス ホメオボックス | ||||||
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / TRANSCRIPTION (転写 (生物学)) /  Structural Genomics (構造ゲノミクス) / NORTHEAST STRUCTURAL GENOMICS CONSORTIUM / NESG / PSI-Biology / Structural Genomics (構造ゲノミクス) / NORTHEAST STRUCTURAL GENOMICS CONSORTIUM / NESG / PSI-Biology /  Protein Structure Initiative / Protein Structure Initiative /  transcription factor (転写因子) / Methods Development transcription factor (転写因子) / Methods Development | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / MADS box domain binding / positive regulation of androgen secretion / regulation of cell cycle => GO:0051726 / salivary gland development / : / dorsal aorta development / : / cellular response to xenobiotic stimulus => GO:0071466 / : ...: / MADS box domain binding / positive regulation of androgen secretion / regulation of cell cycle => GO:0051726 / salivary gland development / : / dorsal aorta development / : / cellular response to xenobiotic stimulus => GO:0071466 / : / negative regulation of insulin-like growth factor receptor signaling pathway / negative regulation of epithelial cell proliferation involved in prostate gland development / multicellular organism development / chromatin => GO:0000785 / epithelial cell proliferation involved in salivary gland morphogenesis / branching involved in prostate gland morphogenesis / pharyngeal system development / metanephros development / cellular response to steroid hormone stimulus / branching morphogenesis of an epithelial tube / response to testosterone / nuclear estrogen receptor activity / androgen receptor signaling pathway / site of DNA damage / cysteine-type endopeptidase activator activity involved in apoptotic process / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process /  transcription factor binding / positive regulation of cell division / negative regulation of mitotic cell cycle / cellular response to interleukin-1 / transcription factor binding / positive regulation of cell division / negative regulation of mitotic cell cycle / cellular response to interleukin-1 /  somitogenesis / positive regulation of intrinsic apoptotic signaling pathway / positive regulation of mitotic cell cycle / phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of apoptotic signaling pathway / nuclear estrogen receptor binding / positive regulation of DNA-binding transcription factor activity / somitogenesis / positive regulation of intrinsic apoptotic signaling pathway / positive regulation of mitotic cell cycle / phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of apoptotic signaling pathway / nuclear estrogen receptor binding / positive regulation of DNA-binding transcription factor activity /  histone deacetylase binding / activation of cysteine-type endopeptidase activity involved in apoptotic process / histone deacetylase binding / activation of cysteine-type endopeptidase activity involved in apoptotic process /  nuclear receptor activity / nuclear receptor activity /  : / : /  regulation of protein localization / negative regulation of epithelial cell proliferation / male gonad development / sequence-specific double-stranded DNA binding / cellular response to tumor necrosis factor / regulation of protein localization / negative regulation of epithelial cell proliferation / male gonad development / sequence-specific double-stranded DNA binding / cellular response to tumor necrosis factor /  heart development / cellular response to hypoxia / sequence-specific DNA binding / heart development / cellular response to hypoxia / sequence-specific DNA binding /  細胞分化 / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / positive regulation of protein phosphorylation / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / negative regulation of cell population proliferation / negative regulation of gene expression / negative regulation of DNA-templated transcription / positive regulation of cell population proliferation / regulation of DNA-templated transcription / positive regulation of gene expression / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / 細胞分化 / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / positive regulation of protein phosphorylation / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / negative regulation of cell population proliferation / negative regulation of gene expression / negative regulation of DNA-templated transcription / positive regulation of cell population proliferation / regulation of DNA-templated transcription / positive regulation of gene expression / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II /  核質 / 核質 /  細胞核 / 細胞核 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  溶液NMR / distance geometry, torsion angle dynamics, 溶液NMR / distance geometry, torsion angle dynamics,  molecular dynamics molecular dynamics | ||||||
| Model details | lowest energy, model 1 | ||||||
 データ登録者 データ登録者 | Liu, G. / Xiao, R. / Lee, H.-W. / Hamilton, K. / Ciccosanti, C. / Wang, H.B. / Acton, T.B. / Everett, J.K. / Huang, Y.J. / Montelione, G.T. / Northeast Structural Genomics Consortium (NESG) | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Solution NMR Structure of Homeobox domain of Homeobox protein Nkx-3.1 from homo sapiens, Northeast Structural Genomics Consortium Target HR6470A 著者: Liu, G. / Xiao, R. / Lee, H.-W. / Hamilton, K. / Ciccosanti, C. / Wang, H.B. / Acton, T.B. / Everett, J.K. / Huang, Y.J. / Montelione, G.T. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2l9r.cif.gz 2l9r.cif.gz | 529.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2l9r.ent.gz pdb2l9r.ent.gz | 453 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2l9r.json.gz 2l9r.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/l9/2l9r https://data.pdbj.org/pub/pdb/validation_reports/l9/2l9r ftp://data.pdbj.org/pub/pdb/validation_reports/l9/2l9r ftp://data.pdbj.org/pub/pdb/validation_reports/l9/2l9r | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
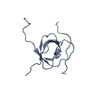
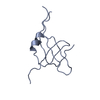
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 |  ホメオボックス / Homeobox protein NK-3 homolog A ホメオボックス / Homeobox protein NK-3 homolog A分子量: 8370.620 Da / 分子数: 1 / 断片: DNA binding Homeobox residues 132-189 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: NKX3-1, NKX3.1, NKX3A / 発現宿主: Homo sapiens (ヒト) / 遺伝子: NKX3-1, NKX3.1, NKX3A / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: Q99801 Escherichia coli (大腸菌) / 参照: UniProt: Q99801 |
|---|
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||
| 試料状態 | pH: 6.5 / 圧: ambient / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: distance geometry, torsion angle dynamics,  molecular dynamics molecular dynamicsソフトェア番号: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj
