+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1x04 | ||||||
|---|---|---|---|---|---|---|---|
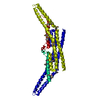
| タイトル | Crystal structure of endophilin BAR domain (mutant) | ||||||
 要素 要素 | SH3-containing GRB2-like protein 2 | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  BAR domain BAR domain | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of blood-brain barrier permeability / dendrite extension / synaptic vesicle uncoating / Retrograde neurotrophin signalling / Lysosome Vesicle Biogenesis / Golgi Associated Vesicle Biogenesis / Recycling pathway of L1 / cellular response to brain-derived neurotrophic factor stimulus / MHC class II antigen presentation / InlB-mediated entry of Listeria monocytogenes into host cell ...negative regulation of blood-brain barrier permeability / dendrite extension / synaptic vesicle uncoating / Retrograde neurotrophin signalling / Lysosome Vesicle Biogenesis / Golgi Associated Vesicle Biogenesis / Recycling pathway of L1 / cellular response to brain-derived neurotrophic factor stimulus / MHC class II antigen presentation / InlB-mediated entry of Listeria monocytogenes into host cell / negative regulation of protein phosphorylation /  central nervous system development / cell projection / EGFR downregulation / clathrin-coated endocytic vesicle membrane / Negative regulation of MET activity / neuron projection development / Cargo recognition for clathrin-mediated endocytosis / presynapse / central nervous system development / cell projection / EGFR downregulation / clathrin-coated endocytic vesicle membrane / Negative regulation of MET activity / neuron projection development / Cargo recognition for clathrin-mediated endocytosis / presynapse /  Clathrin-mediated endocytosis / Clathrin-mediated endocytosis /  エンドソーム / エンドソーム /  ゴルジ体 / negative regulation of gene expression / glutamatergic synapse / ゴルジ体 / negative regulation of gene expression / glutamatergic synapse /  lipid binding / perinuclear region of cytoplasm / lipid binding / perinuclear region of cytoplasm /  シグナル伝達 / identical protein binding / シグナル伝達 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.9 Å 分子置換 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Masuda, M. / Takeda, S. / Sone, M. / Kamioka, Y. / Mori, H. / Mochizuki, N. | ||||||
 引用 引用 |  ジャーナル: Embo J. / 年: 2006 ジャーナル: Embo J. / 年: 2006タイトル: Endophilin BAR domain drives membrane curvature by two newly identified structure-based mechanisms 著者: Masuda, M. / Takeda, S. / Sone, M. / Ohki, T. / Mori, H. / Kamioka, Y. / Mochizuki, N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1x04.cif.gz 1x04.cif.gz | 45.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1x04.ent.gz pdb1x04.ent.gz | 36.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1x04.json.gz 1x04.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/x0/1x04 https://data.pdbj.org/pub/pdb/validation_reports/x0/1x04 ftp://data.pdbj.org/pub/pdb/validation_reports/x0/1x04 ftp://data.pdbj.org/pub/pdb/validation_reports/x0/1x04 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | The biological assembly is a dimer generated by the two fold axis |
- 要素
要素
| #1: タンパク質 | 分子量: 26610.146 Da / 分子数: 1 / 断片: endophilin A1 BAR domain (residues 59-66) / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / プラスミド: pGEX6p3 / 発現宿主: Homo sapiens (ヒト) / プラスミド: pGEX6p3 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): B834(DE3)pLysS Escherichia coli (大腸菌) / 株 (発現宿主): B834(DE3)pLysS参照: UniProt: Q99962,  転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 7.67 Å3/Da / 溶媒含有率: 83.96 % |
|---|---|
結晶化 | 温度: 277 K / 手法: microdialysis / pH: 8 詳細: NaCl, ethylene glycol, glycerol, benzamizine/HCl, Tris, DTT, pH 8.0, MICRODIALYSIS, temperature 277K |
-データ収集
| 回折 | 平均測定温度: 90 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SPring-8 SPring-8  / ビームライン: BL45PX / 波長: 1 Å / ビームライン: BL45PX / 波長: 1 Å |
| 検出器 | タイプ: RIGAKU JUPITER / 検出器: CCD / 日付: 2004年11月27日 |
| 放射 | モノクロメーター: Diamond / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.9→20 Å / Num. all: 17816 / Num. obs: 17780 / % possible obs: 99.8 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Rmerge(I) obs: 0.074 / Net I/σ(I): 20.7 |
| 反射 シェル | 解像度: 2.9→3 Å / Rmerge(I) obs: 0.35 / Mean I/σ(I) obs: 5.5 / Num. unique all: 1782 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 2.9→20 Å / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber 分子置換 / 解像度: 2.9→20 Å / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber
| |||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 74.8 Å2 | |||||||||||||||||||||||||
| Refine analyze |
| |||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.9→20 Å
| |||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.9→3 Å
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj















