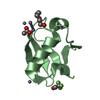
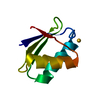
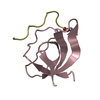
登録情報 データベース : PDB / ID : 1sb6タイトル Solution structure of a cyanobacterial copper metallochaperone, ScAtx1 copper chaperone ScAtx1 キーワード / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / 生物種 Synechocystis sp. (バクテリア)手法 / データ登録者 Banci, L. / Bertini, I. / Ciofi-Baffoni, S. / Su, X.C. / Borrelly, G.P. / Robinson, N.J. / Structural Proteomics in Europe (SPINE) ジャーナル : J.Biol.Chem. / 年 : 2004タイトル : Solution Structures of a Cyanobacterial Metallochaperone: INSIGHT INTO AN ATYPICAL COPPER-BINDING MOTIF.著者 : Banci, L. / Bertini, I. / Ciofi-Baffoni, S. / Su, X.C. / Borrelly, G.P. / Robinson, N.J. 履歴 登録 2004年2月10日 登録サイト / 処理サイト 改定 1.0 2004年4月27日 Provider / タイプ 改定 1.1 2008年4月29日 Group 改定 1.2 2011年7月13日 Group / Version format compliance改定 1.3 2024年5月1日 Group / Database referencesカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード CHAPERONE (シャペロン) / copper chaperone / new metal binding motif /
CHAPERONE (シャペロン) / copper chaperone / new metal binding motif /  Structural Proteomics in Europe (構造ゲノミクス) / SPINE /
Structural Proteomics in Europe (構造ゲノミクス) / SPINE /  Structural Genomics (構造ゲノミクス)
Structural Genomics (構造ゲノミクス) 機能・相同性情報
機能・相同性情報
 溶液NMR / distance geometry,
溶液NMR / distance geometry,  simulated annealing,
simulated annealing,  molecular dynamics, torsion angle dynamics
molecular dynamics, torsion angle dynamics  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2004
ジャーナル: J.Biol.Chem. / 年: 2004 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1sb6.cif.gz
1sb6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1sb6.ent.gz
pdb1sb6.ent.gz PDB形式
PDB形式 1sb6.json.gz
1sb6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/sb/1sb6
https://data.pdbj.org/pub/pdb/validation_reports/sb/1sb6 ftp://data.pdbj.org/pub/pdb/validation_reports/sb/1sb6
ftp://data.pdbj.org/pub/pdb/validation_reports/sb/1sb6 リンク
リンク 集合体
集合体
 要素
要素

 Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P73213
Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P73213 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 simulated annealing,
simulated annealing,  molecular dynamics, torsion angle dynamics
molecular dynamics, torsion angle dynamics ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj
