| 登録情報 | データベース: PDB / ID: 1ksb
|
|---|
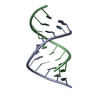
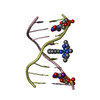
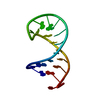
| タイトル | Relationship of Solution and Protein-Bound Structures of DNA Duplexes with the Major Intrastrand Cross-Link Lesions Formed on Cisplatin Binding to DNA |
|---|
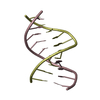
 要素 要素 | - 5'-D(*AP*GP*GP*CP*CP*GP*GP*AP*G)-3'
- 5'-D(*CP*TP*CP*CP*GP*GP*CP*CP*T)-3'
|
|---|
 キーワード キーワード |  DNA (デオキシリボ核酸) / DNA (デオキシリボ核酸) /  Deoxyribonucleic acid (デオキシリボ核酸) / Deoxyribonucleic acid (デオキシリボ核酸) /  Cisplatin (シスプラチン) / Duplex / 9-mer / Intrastrand cross-link / model J Cisplatin (シスプラチン) / Duplex / 9-mer / Intrastrand cross-link / model J |
|---|
| 機能・相同性 |  シスプラチン / シスプラチン /  デオキシリボ核酸 デオキシリボ核酸 機能・相同性情報 機能・相同性情報 |
|---|
| 手法 |  溶液NMR / 溶液NMR /  AMBER AMBER |
|---|
 データ登録者 データ登録者 | Marzilli, L.G. / Saad, J.S. / Kuklenyik, Z. / Keating, K.A. / Xu, Y. |
|---|
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2001 ジャーナル: J.Am.Chem.Soc. / 年: 2001
タイトル: Relationship of solution and protein-bound structures of DNA duplexes with the major intrastrand cross-link lesions formed on cisplatin binding to DNA.
著者: Marzilli, L.G. / Saad, J.S. / Kuklenyik, Z. / Keating, K.A. / Xu, Y. |
|---|
| 履歴 | | 登録 | 2002年1月11日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2002年1月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Non-polymer description / Version format compliance |
|---|
| 改定 1.3 | 2024年5月1日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_software / struct_conn / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード DNA (デオキシリボ核酸) /
DNA (デオキシリボ核酸) /  Deoxyribonucleic acid (デオキシリボ核酸) /
Deoxyribonucleic acid (デオキシリボ核酸) /  Cisplatin (シスプラチン) / Duplex / 9-mer / Intrastrand cross-link / model J
Cisplatin (シスプラチン) / Duplex / 9-mer / Intrastrand cross-link / model J シスプラチン /
シスプラチン /  デオキシリボ核酸
デオキシリボ核酸 機能・相同性情報
機能・相同性情報 溶液NMR /
溶液NMR /  AMBER
AMBER  データ登録者
データ登録者 引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2001
ジャーナル: J.Am.Chem.Soc. / 年: 2001 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1ksb.cif.gz
1ksb.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1ksb.ent.gz
pdb1ksb.ent.gz PDB形式
PDB形式 1ksb.json.gz
1ksb.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ks/1ksb
https://data.pdbj.org/pub/pdb/validation_reports/ks/1ksb ftp://data.pdbj.org/pub/pdb/validation_reports/ks/1ksb
ftp://data.pdbj.org/pub/pdb/validation_reports/ks/1ksb リンク
リンク 集合体
集合体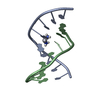
 要素
要素 シスプラチン
シスプラチン 溶液NMR
溶液NMR 試料調製
試料調製
 解析
解析 AMBER / ソフトェア番号: 1
AMBER / ソフトェア番号: 1  ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj



