登録情報 データベース : PDB / ID : 1jm6タイトル Pyruvate dehydrogenase kinase, isozyme 2, containing ADP Pyruvate dehydrogenase kinase, isozyme 2 キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Rattus norvegicus (ドブネズミ)手法 / / / 解像度 : 2.5 Å データ登録者 Steussy, C.N. / Popov, K.M. / Bowker-Kinley, M.M. / Sloan, R.B. / Harris, R.A. / Hamilton, J.A. ジャーナル : J.Biol.Chem. / 年 : 2001タイトル : Structure of pyruvate dehydrogenase kinase. Novel folding pattern for a serine protein kinase.著者 : Steussy, C.N. / Popov, K.M. / Bowker-Kinley, M.M. / Sloan Jr., R.B. / Harris, R.A. / Hamilton, J.A. 履歴 登録 2001年7月17日 登録サイト / 処理サイト 改定 1.0 2001年10月24日 Provider / タイプ 改定 1.1 2008年4月27日 Group 改定 1.2 2011年7月13日 Group 改定 1.3 2024年2月7日 Group / Database references / Derived calculationsカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_struct_conn_angle / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
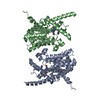
基本情報 要素
要素
 キーワード
キーワード TRANSFERASE (転移酵素) /
TRANSFERASE (転移酵素) /  Kinase (キナーゼ) / Mitochondion /
Kinase (キナーゼ) / Mitochondion /  Serine Kinase
Serine Kinase 機能・相同性情報
機能・相同性情報 ピルビン酸デヒドロゲナーゼ複合体 /
ピルビン酸デヒドロゲナーゼ複合体 /  : / regulation of cellular ketone metabolic process / regulation of pH / cellular response to nutrient ...Signaling by Retinoic Acid / Regulation of pyruvate dehydrogenase (PDH) complex / [pyruvate dehydrogenase (acetyl-transferring)] kinase / regulation of acetyl-CoA biosynthetic process from pyruvate / pyruvate dehydrogenase (acetyl-transferring) kinase activity /
: / regulation of cellular ketone metabolic process / regulation of pH / cellular response to nutrient ...Signaling by Retinoic Acid / Regulation of pyruvate dehydrogenase (PDH) complex / [pyruvate dehydrogenase (acetyl-transferring)] kinase / regulation of acetyl-CoA biosynthetic process from pyruvate / pyruvate dehydrogenase (acetyl-transferring) kinase activity /  ピルビン酸デヒドロゲナーゼ複合体 /
ピルビン酸デヒドロゲナーゼ複合体 /  : / regulation of cellular ketone metabolic process / regulation of pH / cellular response to nutrient /
: / regulation of cellular ketone metabolic process / regulation of pH / cellular response to nutrient /  regulation of gluconeogenesis / intrinsic apoptotic signaling pathway by p53 class mediator / regulation of glucose metabolic process / regulation of calcium-mediated signaling / cellular response to reactive oxygen species / insulin receptor signaling pathway /
regulation of gluconeogenesis / intrinsic apoptotic signaling pathway by p53 class mediator / regulation of glucose metabolic process / regulation of calcium-mediated signaling / cellular response to reactive oxygen species / insulin receptor signaling pathway /  glucose homeostasis / peptidyl-serine phosphorylation / protein serine/threonine kinase activity / protein-containing complex binding / protein homodimerization activity /
glucose homeostasis / peptidyl-serine phosphorylation / protein serine/threonine kinase activity / protein-containing complex binding / protein homodimerization activity /  ミトコンドリア /
ミトコンドリア /  ATP binding / identical protein binding
ATP binding / identical protein binding
 Rattus norvegicus (ドブネズミ)
Rattus norvegicus (ドブネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  AB INITIO / 解像度: 2.5 Å
AB INITIO / 解像度: 2.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2001
ジャーナル: J.Biol.Chem. / 年: 2001 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1jm6.cif.gz
1jm6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1jm6.ent.gz
pdb1jm6.ent.gz PDB形式
PDB形式 1jm6.json.gz
1jm6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/jm/1jm6
https://data.pdbj.org/pub/pdb/validation_reports/jm/1jm6 ftp://data.pdbj.org/pub/pdb/validation_reports/jm/1jm6
ftp://data.pdbj.org/pub/pdb/validation_reports/jm/1jm6 リンク
リンク 集合体
集合体
 要素
要素 / E.C.2.7.1.99 / [PYRUVATE DEHYDROGENASE [LIPOAMIDE]] KINASE ISOZYME 2 / MITOCHONDRIAL / PDK P45
/ E.C.2.7.1.99 / [PYRUVATE DEHYDROGENASE [LIPOAMIDE]] KINASE ISOZYME 2 / MITOCHONDRIAL / PDK P45
 Rattus norvegicus (ドブネズミ) / プラスミド: Pet28a (Novagen) / 発現宿主:
Rattus norvegicus (ドブネズミ) / プラスミド: Pet28a (Novagen) / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21[DE3] / 参照: UniProt: Q64536, EC: 2.7.1.99
Escherichia coli (大腸菌) / 株 (発現宿主): BL21[DE3] / 参照: UniProt: Q64536, EC: 2.7.1.99 アデノシン二リン酸
アデノシン二リン酸 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 解析
解析 :
:  AB INITIO / 解像度: 2.5→6 Å / Num. parameters: 22712 / Num. restraintsaints: 29468 / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER
AB INITIO / 解像度: 2.5→6 Å / Num. parameters: 22712 / Num. restraintsaints: 29468 / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







