+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1j9v | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Solution structure of a lactam analogue (DabD) of HIV gp41 600-612 loop. | ||||||
 要素 要素 | DabD (Ace)IWG(DAB)SGKLIDTTA ANALOGUE OF HIV GP41 | ||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / lactam peptide / lactam bond / VIRAL PROTEIN (ウイルスタンパク質) / lactam peptide / lactam bond /  HIV (ヒト免疫不全ウイルス) / HIV (ヒト免疫不全ウイルス) /  gp41 / gp41 /  cyclic peptide (環状ペプチド) cyclic peptide (環状ペプチド) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Dectin-2 family / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane ...Dectin-2 family / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane ...Dectin-2 family / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity /  生体膜 生体膜類似検索 - 分子機能 | ||||||
| 手法 |  溶液NMR / Torsion angle dinamics, 溶液NMR / Torsion angle dinamics,  molecular dynamics, energy minimization. molecular dynamics, energy minimization. | ||||||
 データ登録者 データ登録者 | Phan Chan Du, A. / Limal, D. / Semetey, V. / Dali, H. / Jolivet, M. / Desgranges, C. / Cung, M.T. / Briand, J.P. / Petit, M.C. / Muller, S. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2002 ジャーナル: J.Mol.Biol. / 年: 2002タイトル: Structural and immunological characterisation of heteroclitic peptide analogues corresponding to the 600-612 region of the HIV envelope gp41 glycoprotein. 著者: Du, A.P. / Limal, D. / Semetey, V. / Dali, H. / Jolivet, M. / Desgranges, C. / Cung, M.T. / Briand, J.P. / Petit, M.C. / Muller, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1j9v.cif.gz 1j9v.cif.gz | 164.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1j9v.ent.gz pdb1j9v.ent.gz | 123.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1j9v.json.gz 1j9v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/j9/1j9v https://data.pdbj.org/pub/pdb/validation_reports/j9/1j9v ftp://data.pdbj.org/pub/pdb/validation_reports/j9/1j9v ftp://data.pdbj.org/pub/pdb/validation_reports/j9/1j9v | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 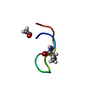
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 1388.589 Da / 分子数: 1 / 由来タイプ: 合成 詳細: The parent peptide (IWGCSGKLICTTA) occurs in HIV gp41 glycoprotein. 参照: UniProt: P12488 |
|---|
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||
| NMR実験の詳細 | Text: This structure was determined using standard 2D homonuclear techniques. Different NOESY experiments with mixing times from 80 ms to 800 ms were recorded, in order to determined the best ...Text: This structure was determined using standard 2D homonuclear techniques. Different NOESY experiments with mixing times from 80 ms to 800 ms were recorded, in order to determined the best conditions avoiding spin diffusion. |
- 試料調製
試料調製
| 詳細 | 内容: 3,5 mM peptide in 500ul DMSO-D6 / 溶媒系: DMSO-D6 |
|---|---|
| 試料状態 | 圧: ambient / 温度: 298 K |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射波長 | 相対比: 1 | |||||||||||||||
| NMRスペクトロメーター |
|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: Torsion angle dinamics,  molecular dynamics, energy minimization. molecular dynamics, energy minimization.ソフトェア番号: 1 詳細: 50 INITIAL RANDOM STRUCTURES WERE GENERATED USING SIMULATED ANNEALING IN DYANA SOFTWARE, FOLLOWED BY 500PS RESTRAINED MINIMIZATION, 35PS MD IN VACUO AT 300K, 200 PS MD UNDER NMR RESTRAINTS ...詳細: 50 INITIAL RANDOM STRUCTURES WERE GENERATED USING SIMULATED ANNEALING IN DYANA SOFTWARE, FOLLOWED BY 500PS RESTRAINED MINIMIZATION, 35PS MD IN VACUO AT 300K, 200 PS MD UNDER NMR RESTRAINTS AND 750 STEPS CONJUGATED GRADIENT EM USING DISCOVER OF MSI SOFTWARE | ||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 49 |
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj




