+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1hfe | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | 1.6 A RESOLUTION STRUCTURE OF THE FE-ONLY HYDROGENASE FROM DESULFOVIBRIO DESULFURICANS | ||||||
 要素 要素 | (PROTEIN (FE-ONLY HYDROGENASE (E.C.1.18.99.1) ...) x 2 | ||||||
 キーワード キーワード |  HYDROGENASE (ヒドロゲナーゼ) / FE-ONLY HYDROGENASE / HYDROGENE METABOLISM / HYDROGENASE (ヒドロゲナーゼ) / FE-ONLY HYDROGENASE / HYDROGENE METABOLISM /  PERIPLASM (ペリプラズム) PERIPLASM (ペリプラズム) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 フェレドキシンヒドロゲナーゼ / フェレドキシンヒドロゲナーゼ /  ferredoxin hydrogenase activity / ferredoxin hydrogenase activity /  iron-sulfur cluster binding / 4 iron, 4 sulfur cluster binding / iron-sulfur cluster binding / 4 iron, 4 sulfur cluster binding /  ペリプラズム / ペリプラズム /  electron transfer activity / iron ion binding electron transfer activity / iron ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Desulfovibrio vulgaris subsp. vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris subsp. vulgaris str. Hildenborough (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 1.6 Å 多波長異常分散 / 解像度: 1.6 Å | ||||||
 データ登録者 データ登録者 | Nicolet, Y. / Piras, C. / Legrand, P. / Hatchikian, E.C. / Fontecilla-Camps, J.C. | ||||||
 引用 引用 |  ジャーナル: Structure Fold.Des. / 年: 1999 ジャーナル: Structure Fold.Des. / 年: 1999タイトル: Desulfovibrio desulfuricans iron hydrogenase: the structure shows unusual coordination to an active site Fe binuclear center. 著者: Nicolet, Y. / Piras, C. / Legrand, P. / Hatchikian, C.E. / Fontecilla-Camps, J.C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1hfe.cif.gz 1hfe.cif.gz | 238.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1hfe.ent.gz pdb1hfe.ent.gz | 185.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1hfe.json.gz 1hfe.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hf/1hfe https://data.pdbj.org/pub/pdb/validation_reports/hf/1hfe ftp://data.pdbj.org/pub/pdb/validation_reports/hf/1hfe ftp://data.pdbj.org/pub/pdb/validation_reports/hf/1hfe | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-PROTEIN (FE-ONLY HYDROGENASE (E.C.1.18.99.1) ... , 2種, 4分子 STLM
| #1: タンパク質 | 分子量: 13646.787 Da / 分子数: 2 / 由来タイプ: 天然 詳細: THE FE-ONLY HYDROGENASE FROM DESULFOVIBRIO DESULFURICANS HAS EXACTLY THE SAME SEQUENCE AS THE FE-ONLY HYDROGENASE FROM DESULFOVIBRIO VULGARIS (STRAIN HILDENBOROUGH) 由来: (天然)   Desulfovibrio vulgaris subsp. vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris subsp. vulgaris str. Hildenborough (バクテリア)細胞内の位置: PERIPLASM  ペリプラズム / 生物種: Desulfovibrio vulgaris ペリプラズム / 生物種: Desulfovibrio vulgaris / 株: Hildenborough / ATCC 29579 / NCIMB 8303 / 参照: UniProt: P07603, 1.18.99.1 / 株: Hildenborough / ATCC 29579 / NCIMB 8303 / 参照: UniProt: P07603, 1.18.99.1#2: タンパク質 | 分子量: 46011.133 Da / 分子数: 2 / 由来タイプ: 天然 詳細: THE FE-ONLY HYDROGENASE FROM DESULFOVIBRIO DESULFURICANS HAS EXACTLY THE SAME SEQUENCE AS THE FE-ONLY HYDROGENASE FROM DESULFOVIBRIO VULGARIS (STRAIN HILDENBOROUGH) 由来: (天然)   Desulfovibrio vulgaris subsp. vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris subsp. vulgaris str. Hildenborough (バクテリア)細胞内の位置: PERIPLASM  ペリプラズム / 生物種: Desulfovibrio vulgaris ペリプラズム / 生物種: Desulfovibrio vulgaris / 株: Hildenborough / ATCC 29579 / NCIMB 8303 / 参照: UniProt: P07598, 1.18.99.1 / 株: Hildenborough / ATCC 29579 / NCIMB 8303 / 参照: UniProt: P07598, 1.18.99.1 |
|---|
-非ポリマー , 8種, 1202分子 



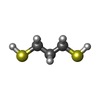

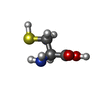




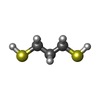



| #3: 化合物 | ChemComp-ZN / | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| #4: 化合物 | ChemComp-FE2 / #5: 化合物 | ChemComp-CYN /  シアン化物 シアン化物#6: 化合物 | ChemComp-SF4 /  鉄・硫黄クラスター 鉄・硫黄クラスター#7: 化合物 |  Propane-1,3-dithiol Propane-1,3-dithiol#8: 化合物 | ChemComp-CMO /  一酸化炭素 一酸化炭素#9: 化合物 |  システイン システイン#10: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.32 Å3/Da / 溶媒含有率: 35 % | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | pH: 7.6 / 詳細: pH 7.60 | ||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5 | ||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID14-3 / 波長: 0.947375 / ビームライン: ID14-3 / 波長: 0.947375 |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 1998年5月1日 / 詳細: MIRROR |
| 放射 | モノクロメーター: SI(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.947375 Å / 相対比: 1 : 0.947375 Å / 相対比: 1 |
| 反射 | 解像度: 1.51→21 Å / Num. obs: 158363 / % possible obs: 94.3 % / 冗長度: 6.4 % / Biso Wilson estimate: 14.27 Å2 / Rsym value: 0.071 / Net I/σ(I): 4.8 |
| 反射 シェル | 解像度: 1.51→1.6 Å / 冗長度: 5.5 % / Mean I/σ(I) obs: 1.1 / Rsym value: 0.452 / % possible all: 89.2 |
| 反射 | *PLUS Rmerge(I) obs: 0.071 |
| 反射 シェル | *PLUS % possible obs: 89.2 % / Rmerge(I) obs: 0.452 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  多波長異常分散 / 解像度: 1.6→7 Å / Rfactor Rfree error: 0.002 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 2 多波長異常分散 / 解像度: 1.6→7 Å / Rfactor Rfree error: 0.002 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 2 詳細: THE 2 MOLECULES OF THE ASYMMETRIC UNIT WERE NEVER CONSTRAINT WITH THE NON CRYSTALLOGRAPHIC SYMMETRY DURING REFINEMENT.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 16.52 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.6→7 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.6→1.67 Å / Rfactor Rfree error: 0.01 / Total num. of bins used: 8
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.8 / 分類: refinement X-PLOR / バージョン: 3.8 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


























