+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1aqz | ||||||
|---|---|---|---|---|---|---|---|
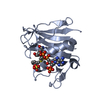
| タイトル | CRYSTAL STRUCTURE OF A HIGHLY SPECIFIC ASPERGILLUS RIBOTOXIN, RESTRICTOCIN | ||||||
 要素 要素 | RESTRICTOCIN | ||||||
 キーワード キーワード | RIBOTOXIN /  RIBOSOME-INACTIVATING PROTEIN / PROTEIN-RNA SPECIFIC INTERACTION / RIBOSOME-INACTIVATING PROTEIN / PROTEIN-RNA SPECIFIC INTERACTION /  LAUE DIFFRACTION (X線回折) / CELL-ENTRY ACTIVITY LAUE DIFFRACTION (X線回折) / CELL-ENTRY ACTIVITY | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ / RNA nuclease activity / negative regulation of translation / 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ / RNA nuclease activity / negative regulation of translation /  RNA binding / extracellular region RNA binding / extracellular region類似検索 - 分子機能 | ||||||
| 生物種 |   Aspergillus restrictus (カビ) Aspergillus restrictus (カビ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / SINGLE ISOMORPHOUS REPLACEMENT, シンクロトロン / SINGLE ISOMORPHOUS REPLACEMENT,  ANOMALOUS SCATTERING / 解像度: 1.7 Å ANOMALOUS SCATTERING / 解像度: 1.7 Å | ||||||
 データ登録者 データ登録者 | Yang, X. / Moffat, K. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 1996 ジャーナル: Structure / 年: 1996タイトル: Insights into specificity of cleavage and mechanism of cell entry from the crystal structure of the highly specific Aspergillus ribotoxin, restrictocin. 著者: Yang, X. / Moffat, K. #1:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1993 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1993タイトル: The Conformation of the Sarcin/Ricin Loop from 28S Ribosomal RNA 著者: Szewczak, A.A. / Moore, P.B. / Chan, Y.L. / Wool, I.G. #2:  ジャーナル: Acta Crystallogr.,Sect.B / 年: 1991 ジャーナル: Acta Crystallogr.,Sect.B / 年: 1991タイトル: Determination and Restrained Least-Squares Refinement of the Structures of Ribonuclease Sa and its Complex with 3'-Guanylic Acid at 1.8 A Resolution 著者: Sevcik, J. / Dodson, E.J. / Dodson, G.G. #3:  ジャーナル: J.Mol.Biol. / 年: 1991 ジャーナル: J.Mol.Biol. / 年: 1991タイトル: Crystallization and Preliminary Characterization of Mitogillin, a Ribosomal Ribonuclease from Aspergillus Restrictus 著者: Martinez, S.E. / Smith, J.L. #4:  ジャーナル: Trends Biochem.Sci. / 年: 1984 ジャーナル: Trends Biochem.Sci. / 年: 1984タイトル: The Mechanism of Action of the Cytotoxic Nuclease Alpha-Sarcin and its Use to Analyse Ribosome Structure 著者: Wool, I.G. #5:  ジャーナル: Nature / 年: 1982 ジャーナル: Nature / 年: 1982タイトル: Specific Protein-Nucleic Acid Recognition in Ribonuclease T1-2'-Guanylic Acid Complex: An X-Ray Study 著者: Heinemann, U. / Saenger, W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1aqz.cif.gz 1aqz.cif.gz | 69.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1aqz.ent.gz pdb1aqz.ent.gz | 56.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1aqz.json.gz 1aqz.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/aq/1aqz https://data.pdbj.org/pub/pdb/validation_reports/aq/1aqz ftp://data.pdbj.org/pub/pdb/validation_reports/aq/1aqz ftp://data.pdbj.org/pub/pdb/validation_reports/aq/1aqz | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (-0.996739, 0.022599, -0.077458), ベクター  : : |
- 要素
要素
| #1: タンパク質 | 分子量: 16888.893 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: INORGANIC PHOSPHATE GROUP AT THE ACTIVE SITE / 由来: (天然)   Aspergillus restrictus (カビ) Aspergillus restrictus (カビ)参照: UniProt: P67876,  加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ#2: 化合物 |  リン酸塩 リン酸塩#3: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.29 Å3/Da / 溶媒含有率: 46 % 解説: DATA WERE COLLECTED USING LAUE DIFFRACTION. 62 IMAGES WERE COLLECTED. TOTAL EXPOSURE TIME IS 325 MS. | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 手法: 蒸気拡散法, microdialysis / pH: 6.8 詳細: A COMBINATION OF VAPOR DIFFUSION AND MICRODIALYSIS TECHNIQUES WERE USED TO CRYSTALLIZE RESTRICTOCIN. THE FINAL MOTHER LIQUOR CONSISTS OF 60% ETHANOL, 10MM SODIUM PHOSPHATE, PH6.8. THE PROTEIN ...詳細: A COMBINATION OF VAPOR DIFFUSION AND MICRODIALYSIS TECHNIQUES WERE USED TO CRYSTALLIZE RESTRICTOCIN. THE FINAL MOTHER LIQUOR CONSISTS OF 60% ETHANOL, 10MM SODIUM PHOSPHATE, PH6.8. THE PROTEIN CONCENTRATION IS 10MG/ML., vapor diffusion and microdialysis | ||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法 | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 298 K | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS NSLS  / ビームライン: X26C / 波長: 0.7 / 波長: 0.7, 2.05 / ビームライン: X26C / 波長: 0.7 / 波長: 0.7, 2.05 | |||||||||
| 検出器 | タイプ: FUJI / 検出器: IMAGE PLATE / 日付: 1994年8月1日 / 詳細: MIRROR | |||||||||
| 放射 | 単色(M)・ラウエ(L): L / 散乱光タイプ: x-ray | |||||||||
| 放射波長 |
| |||||||||
| 反射 | 解像度: 1.7→8 Å / Num. obs: 28328 / % possible obs: 85.6 % / Observed criterion σ(I): 2 / 冗長度: 8 % / Rmerge(I) obs: 0.039 | |||||||||
| 反射 シェル | 解像度: 1.7→1.78 Å / % possible all: 54.8 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : SINGLE ISOMORPHOUS REPLACEMENT, : SINGLE ISOMORPHOUS REPLACEMENT,  ANOMALOUS SCATTERING ANOMALOUS SCATTERING解像度: 1.7→8 Å / 交差検証法: THROUGHOUT / σ(F): 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 19.9 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→8 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.7→1.78 Å / Total num. of bins used: 8
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.1 / 分類: refinement X-PLOR / バージョン: 3.1 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.177 / Rfactor Rfree : 0.237 : 0.237 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj


