+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
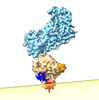
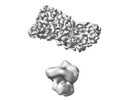
| タイトル | Cryo-EM of the GDP-bound human dynamin polymer assembled on the membrane in the super constricted state (full helix) | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  dynamin / dynamin /  membrane (生体膜) / fission / membrane (生体膜) / fission /  lipid (脂質) / lipid (脂質) /  tubule / scission / tubule / scission /  endocytosis (エンドサイトーシス) / endocytosis (エンドサイトーシス) /  HYDROLASE (加水分解酵素) HYDROLASE (加水分解酵素) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報clathrin coat assembly involved in endocytosis / vesicle scission / synaptic vesicle budding from presynaptic endocytic zone membrane / presynaptic endocytic zone membrane /  dynamin GTPase / dynamin GTPase /  クロム親和性細胞 / regulation of vesicle size / Toll Like Receptor 4 (TLR4) Cascade / Retrograde neurotrophin signalling / Formation of annular gap junctions ...clathrin coat assembly involved in endocytosis / vesicle scission / synaptic vesicle budding from presynaptic endocytic zone membrane / presynaptic endocytic zone membrane / クロム親和性細胞 / regulation of vesicle size / Toll Like Receptor 4 (TLR4) Cascade / Retrograde neurotrophin signalling / Formation of annular gap junctions ...clathrin coat assembly involved in endocytosis / vesicle scission / synaptic vesicle budding from presynaptic endocytic zone membrane / presynaptic endocytic zone membrane /  dynamin GTPase / dynamin GTPase /  クロム親和性細胞 / regulation of vesicle size / Toll Like Receptor 4 (TLR4) Cascade / Retrograde neurotrophin signalling / Formation of annular gap junctions / endosome organization / Gap junction degradation / photoreceptor ribbon synapse / membrane coat / Recycling pathway of L1 / phosphatidylinositol-3,4,5-trisphosphate binding / endocytic vesicle / EPH-ephrin mediated repulsion of cells / クロム親和性細胞 / regulation of vesicle size / Toll Like Receptor 4 (TLR4) Cascade / Retrograde neurotrophin signalling / Formation of annular gap junctions / endosome organization / Gap junction degradation / photoreceptor ribbon synapse / membrane coat / Recycling pathway of L1 / phosphatidylinositol-3,4,5-trisphosphate binding / endocytic vesicle / EPH-ephrin mediated repulsion of cells /  クラスリン / クラスリン /  phosphatidylinositol-4,5-bisphosphate binding / MHC class II antigen presentation / photoreceptor inner segment / phosphatidylinositol-4,5-bisphosphate binding / MHC class II antigen presentation / photoreceptor inner segment /  receptor-mediated endocytosis / cell projection / modulation of chemical synaptic transmission / protein homooligomerization / receptor-mediated endocytosis / cell projection / modulation of chemical synaptic transmission / protein homooligomerization /  receptor internalization / receptor internalization /  エンドサイトーシス / GDP binding / : / presynapse / エンドサイトーシス / GDP binding / : / presynapse /  Clathrin-mediated endocytosis / Clathrin-mediated endocytosis /  microtubule binding / protein homotetramerization / microtubule binding / protein homotetramerization /  微小管 / 微小管 /  GTPase activity / glutamatergic synapse / GTPase activity / glutamatergic synapse /  シナプス / GTP binding / シナプス / GTP binding /  protein kinase binding / protein homodimerization activity / protein kinase binding / protein homodimerization activity /  RNA binding / extracellular exosome / identical protein binding / RNA binding / extracellular exosome / identical protein binding /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.55 Å クライオ電子顕微鏡法 / 解像度: 3.55 Å | ||||||||||||
 データ登録者 データ登録者 | Jimah JR / Canagarajah BJ / Hinshaw JE | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Dev Cell / 年: 2024 ジャーナル: Dev Cell / 年: 2024タイトル: Cryo-EM structures of membrane-bound dynamin in a post-hydrolysis state primed for membrane fission. 著者: John R Jimah / Nidhi Kundu / Abigail E Stanton / Kem A Sochacki / Bertram Canagarajah / Lieza Chan / Marie-Paule Strub / Huaibin Wang / Justin W Taraska / Jenny E Hinshaw /  要旨: Dynamin assembles as a helical polymer at the neck of budding endocytic vesicles, constricting the underlying membrane as it progresses through the GTPase cycle to sever vesicles from the ...Dynamin assembles as a helical polymer at the neck of budding endocytic vesicles, constricting the underlying membrane as it progresses through the GTPase cycle to sever vesicles from the plasma membrane. Although atomic models of the dynamin helical polymer bound to guanosine triphosphate (GTP) analogs define earlier stages of membrane constriction, there are no atomic models of the assembled state post-GTP hydrolysis. Here, we used cryo-EM methods to determine atomic structures of the dynamin helical polymer assembled on lipid tubules, akin to necks of budding endocytic vesicles, in a guanosine diphosphate (GDP)-bound, super-constricted state. In this state, dynamin is assembled as a 2-start helix with an inner lumen of 3.4 nm, primed for spontaneous fission. Additionally, by cryo-electron tomography, we trapped dynamin helical assemblies within HeLa cells using the GTPase-defective dynamin K44A mutant and observed diverse dynamin helices, demonstrating that dynamin can accommodate a range of assembled complexes in cells that likely precede membrane fission. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40903.map.gz emd_40903.map.gz | 57.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40903-v30.xml emd-40903-v30.xml emd-40903.xml emd-40903.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40903_fsc.xml emd_40903_fsc.xml | 18.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40903.png emd_40903.png | 99.9 KB | ||
| マスクデータ |  emd_40903_msk_1.map emd_40903_msk_1.map | 476.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40903.cif.gz emd-40903.cif.gz | 6 KB | ||
| その他 |  emd_40903_half_map_1.map.gz emd_40903_half_map_1.map.gz emd_40903_half_map_2.map.gz emd_40903_half_map_2.map.gz | 442 MB 442 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40903 http://ftp.pdbj.org/pub/emdb/structures/EMD-40903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40903 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8sz8MC  8sxzC  8sz4C  8sz7C  8t0kC  8t0rC  8tymC  8tynC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40903.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40903.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.289 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40903_msk_1.map emd_40903_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40903_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40903_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM of the GDP-bound human dynamin polymer assembled on the m...
| 全体 | 名称: Cryo-EM of the GDP-bound human dynamin polymer assembled on the membrane in the super constricted state (full helix) |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM of the GDP-bound human dynamin polymer assembled on the m...
| 超分子 | 名称: Cryo-EM of the GDP-bound human dynamin polymer assembled on the membrane in the super constricted state (full helix) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 85.7 kDa/nm |
-分子 #1: Dynamin-1
| 分子 | 名称: Dynamin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  dynamin GTPase dynamin GTPase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 86.312625 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: GPTGMGNRGM EDLIPLVNRL QDAFSAIGQN ADLDLPQIAV VGGQSAGASS VLENFVGRDF LPRGSGIVTR RPLVLQLVNA TTEYAEFLH CKGKKFTDFE EVRLEIEAET DRVTGTNKGI SPVPINLRVY SPHVLNLTLV DLPGMTKVPV GDQPPDIEFQ I RDMLMQFV ...文字列: GPTGMGNRGM EDLIPLVNRL QDAFSAIGQN ADLDLPQIAV VGGQSAGASS VLENFVGRDF LPRGSGIVTR RPLVLQLVNA TTEYAEFLH CKGKKFTDFE EVRLEIEAET DRVTGTNKGI SPVPINLRVY SPHVLNLTLV DLPGMTKVPV GDQPPDIEFQ I RDMLMQFV TKENCLILAV SPANSDLANS DALKVAKEVD PQGQRTIGVI TKLDLMDEGT DARDVLENKL LPLRRGYIGV VN RSQKDID GKKDITAALA AERKFFLSHP SYRHLADRMG TPYLQKVLNQ QLTNHIRDTL PGLRNKLQSQ LLSIEKEVEE YKN FRPDDP ARKTKALLQM VQQFAVDFEK RIEGSGDQID TYELSGGARI NRIFHERFPF ELVKMEFDEK ELRREISYAI KNIH GIRTG LFTPDMAFET IVKKQVKKIR EPCLKCVDMV ISELISTVRQ CTKKLQQYPR LREEMERIVT THIREREGRT KEQVM LLID IELAYMNTNH EDFIGFANAQ QRSNQMNKKK TSGNQDEILV IRKGWLTINN IGIMKGGSKE YWFVLTAENL SWYKDD EEK EKKYMLSVDN LKLRDVEKGF MSSKHIFALF NTEQRNVYKD YRQLELACET QEEVDSWKAS FLRAGVYPER VGDKEKA SE TEENGSDSFM HSMDPQLERQ VETIRNLVDS YMAIVNKTVR DLMPKTIMHL MINNTKEFIF SELLANLYSC GDQNTLME E SAEQAQRRDE MLRMYHALKE ALSIIGNINT TTV UniProtKB:  Dynamin-1 Dynamin-1 |
-分子 #2: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.6 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X