+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of RCD-1 pore from Neurospora crassa | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Pyroptosis / Gasdermnin / Pore / Pyroptosis / Gasdermnin / Pore /  Allorecognition / Allorecognition /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||
| 機能・相同性 | wide pore channel activity /  プログラム細胞死 / protein heterodimerization activity / プログラム細胞死 / protein heterodimerization activity /  細胞膜 / 細胞膜 /  細胞質 / Gasdermin-like protein rcd-1-2 / Gasdermin-like protein rcd-1-1 細胞質 / Gasdermin-like protein rcd-1-2 / Gasdermin-like protein rcd-1-1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Neurospora crassa (アカパンカビ) Neurospora crassa (アカパンカビ) | |||||||||
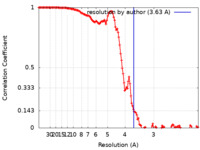
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.63 Å クライオ電子顕微鏡法 / 解像度: 3.63 Å | |||||||||
 データ登録者 データ登録者 | Hou YJ / Sun Q / Li Y / Ding J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Cleavage-independent activation of ancient eukaryotic gasdermins and structural mechanisms. 著者: Yueyue Li / Yanjie Hou / Qi Sun / Huan Zeng / Fanyi Meng / Xiang Tian / Qun He / Feng Shao / Jingjin Ding /  要旨: Gasdermins (GSDMs) are pore-forming proteins that execute pyroptosis for immune defense. GSDMs are two-domain proteins activated by proteolytic removal of the inhibitory domain. In this work, we ...Gasdermins (GSDMs) are pore-forming proteins that execute pyroptosis for immune defense. GSDMs are two-domain proteins activated by proteolytic removal of the inhibitory domain. In this work, we report two types of cleavage-independent GSDM activation. First, GSDM, a pore-forming domain-only protein from the basal metazoan , is a disulfides-linked autoinhibited dimer activated by reduction of the disulfides. The cryo-electron microscopy (cryo-EM) structure illustrates the assembly mechanism for the 44-mer GSDM pore. Second, RCD-1-1 and RCD-1-2, encoded by the polymorphic () gene in filamentous fungus , are also pore-forming domain-only GSDMs. RCD-1-1 and RCD-1-2, when encountering each other, form pores and cause pyroptosis, underlying allorecognition in . The cryo-EM structure reveals a pore of 11 RCD-1-1/RCD-1-2 heterodimers and a heterodimerization-triggered pore assembly mechanism. This study shows mechanistic diversities in GSDM activation and indicates versatile functions of GSDMs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36734.map.gz emd_36734.map.gz | 197.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36734-v30.xml emd-36734-v30.xml emd-36734.xml emd-36734.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36734_fsc.xml emd_36734_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36734.png emd_36734.png | 178.8 KB | ||
| Filedesc metadata |  emd-36734.cif.gz emd-36734.cif.gz | 5.8 KB | ||
| その他 |  emd_36734_half_map_1.map.gz emd_36734_half_map_1.map.gz emd_36734_half_map_2.map.gz emd_36734_half_map_2.map.gz | 193.5 MB 193.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36734 http://ftp.pdbj.org/pub/emdb/structures/EMD-36734 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36734 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36734 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36734.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36734.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_36734_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
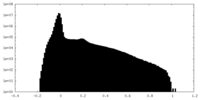
| 投影像・断面図 |
| ||||||||||||
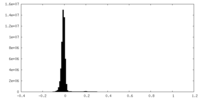
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36734_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
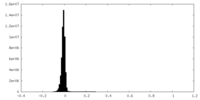
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RCD-1 pore from Neurospora crassa
| 全体 | 名称: RCD-1 pore from Neurospora crassa |
|---|---|
| 要素 |
|
-超分子 #1: RCD-1 pore from Neurospora crassa
| 超分子 | 名称: RCD-1 pore from Neurospora crassa / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Neurospora crassa (アカパンカビ) Neurospora crassa (アカパンカビ) |
| 分子量 | 理論値: 620 KDa |
-分子 #1: Gasdermin-like protein rcd-1-1
| 分子 | 名称: Gasdermin-like protein rcd-1-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Neurospora crassa (アカパンカビ) Neurospora crassa (アカパンカビ) |
| 分子量 | 理論値: 29.218693 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: SGRPMDKCWF TLDNAHYPPP SLDSMRSGHP ISPASLGHLI PSLAHLDQII NAKAIEPFPA TMDIHGPTII EDFKWDHSHE YSLSLGGKV PIPLAPAGVP FVDLNVGLGG AFSRSVANYW EFDRLERYIM QPTRSYVQKC IERDEVKRWI AKNKSMMMMG R WEVYMITG ...文字列: SGRPMDKCWF TLDNAHYPPP SLDSMRSGHP ISPASLGHLI PSLAHLDQII NAKAIEPFPA TMDIHGPTII EDFKWDHSHE YSLSLGGKV PIPLAPAGVP FVDLNVGLGG AFSRSVANYW EFDRLERYIM QPTRSYVQKC IERDEVKRWI AKNKSMMMMG R WEVYMITG IIVARGGGRK KKEKTTGKEF SVEVTVEVPL IVEAGPGGKR NTARQKTWGT SQTGDFVWAV RLAKITKSGL HS DWKMETV FGKTSSFRGQ KAIF UniProtKB: Gasdermin-like protein rcd-1-1 |
-分子 #2: Gasdermin-like protein rcd-1-2
| 分子 | 名称: Gasdermin-like protein rcd-1-2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Neurospora crassa (アカパンカビ) Neurospora crassa (アカパンカビ) |
| 分子量 | 理論値: 27.630418 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: SGRPMDNEEW FPLKQTHYPP PTIPSMKTGH PTGPISIGHI IPDLRHLDNV INCKGFEPFP PNMDVFTAHY EQCHFGDHLN SEFVVQAKA AAPIENIVPG VDVTGSAGLH HTNITSDRWE YDSVVEYAVY PTRQYIDRLL ESKEVRQYIQ KSKKLLGGWC V YMVTGIMV ...文字列: SGRPMDNEEW FPLKQTHYPP PTIPSMKTGH PTGPISIGHI IPDLRHLDNV INCKGFEPFP PNMDVFTAHY EQCHFGDHLN SEFVVQAKA AAPIENIVPG VDVTGSAGLH HTNITSDRWE YDSVVEYAVY PTRQYIDRLL ESKEVRQYIQ KSKKLLGGWC V YMVTGIMV ARGGGRNVTS EEKGAGVSGN VGFQVPGIGE FAPEVGWDTK TKTKVNAHHT TDFVCAIRLV KIAKSGLRSS WT MKKVTRE F UniProtKB: Gasdermin-like protein rcd-1-2 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 56.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X