+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cocktail of FMDV (A/TUR/14/98) in complex with M678F and M688F | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  FMDV (口蹄疫ウイルス) / FMDV (口蹄疫ウイルス) /  antibody (抗体) / antibody (抗体) /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報icosahedral viral capsid / host cell cytoplasm / symbiont entry into host cell / virion attachment to host cell / structural molecule activity /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Foot-and-mouth disease virus A (口蹄疫ウイルス) / Foot-and-mouth disease virus A (口蹄疫ウイルス) /   Lama glama (ラマ) Lama glama (ラマ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Li HZ / Dong H / Liu P | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural and in vivo studies of neutralizing antibody topographical classifications reveal mechanisms underlying differences in immunogenicity and antigenicity between 146S and 12S of ...タイトル: Structural and in vivo studies of neutralizing antibody topographical classifications reveal mechanisms underlying differences in immunogenicity and antigenicity between 146S and 12S of foot-and-mouth disease virus 著者: Li HZ / Dong H / Liu P | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34637.map.gz emd_34637.map.gz | 49.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34637-v30.xml emd-34637-v30.xml emd-34637.xml emd-34637.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34637.png emd_34637.png | 70.2 KB | ||
| Filedesc metadata |  emd-34637.cif.gz emd-34637.cif.gz | 5.7 KB | ||
| その他 |  emd_34637_half_map_1.map.gz emd_34637_half_map_1.map.gz emd_34637_half_map_2.map.gz emd_34637_half_map_2.map.gz | 75.6 MB 75.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34637 http://ftp.pdbj.org/pub/emdb/structures/EMD-34637 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34637 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34637 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34637.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34637.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.36 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_34637_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
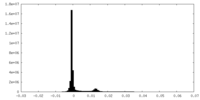
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34637_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : FMDV capsid protein in complex with M678F nab and M688F nab
| 全体 | 名称: FMDV capsid protein in complex with M678F nab and M688F nab |
|---|---|
| 要素 |
|
-超分子 #1: FMDV capsid protein in complex with M678F nab and M688F nab
| 超分子 | 名称: FMDV capsid protein in complex with M678F nab and M688F nab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus A (口蹄疫ウイルス) Foot-and-mouth disease virus A (口蹄疫ウイルス) |
-分子 #1: M678F nab
| 分子 | 名称: M678F nab / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lama glama (ラマ) Lama glama (ラマ) |
| 分子量 | 理論値: 12.753181 KDa |
| 組換発現 | 生物種: Yeast centromeric expression vector p416GPD (その他) |
| 配列 | 文字列: QVQLQESGGG LVQAGGSLRL SCVYSGGAYS MGWYRQAPGK QRELVAAITD DGITNYRDTV KGRFTISRDN AKKAVYLQMN SLKPEDTAV YHCNTVRRVA TLSGSSSGSW GQGTQVTVSS |
-分子 #2: M688F nab
| 分子 | 名称: M688F nab / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lama glama (ラマ) Lama glama (ラマ) |
| 分子量 | 理論値: 13.861437 KDa |
| 組換発現 | 生物種: Yeast centromeric expression vector p416GPD (その他) |
| 配列 | 文字列: QVQLQESGGG LVQAGDSLRL SCVPSVRTSD NYIMGWFRQP PGKEREFVAA IRRSDGTTKY AASVKGRFAI SRDVAKNAAY LQMNSLKAE DTAVYYCAAK YQSTFYSTMD VQYDYWGQGT QVTVSS |
-分子 #3: VP1 of capsid protein
| 分子 | 名称: VP1 of capsid protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus A (口蹄疫ウイルス) Foot-and-mouth disease virus A (口蹄疫ウイルス) |
| 分子量 | 理論値: 23.111094 KDa |
| 配列 | 文字列: TTSAGESADP VTTTVENYGG ETQVQRRHHT DVGFIMDRFV KINNTNPTHV IDLMQTHQHG LVGALLRAAT YYFSDLEIVV RHEGNLTWV PNGAPEAALS NAGNPTAYNK APFTRLALPY TAPHRVLATV YNGTSKYSTT GERTRGDLGA LAARVATQLP A SFNFGAIR ...文字列: TTSAGESADP VTTTVENYGG ETQVQRRHHT DVGFIMDRFV KINNTNPTHV IDLMQTHQHG LVGALLRAAT YYFSDLEIVV RHEGNLTWV PNGAPEAALS NAGNPTAYNK APFTRLALPY TAPHRVLATV YNGTSKYSTT GERTRGDLGA LAARVATQLP A SFNFGAIR ATDISELLVR MKRAELYCPR PLLAVEVTAQ DRHKQKIIAP AKQ UniProtKB: Genome polyprotein |
-分子 #4: VP2 of capsid protein
| 分子 | 名称: VP2 of capsid protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus A (口蹄疫ウイルス) Foot-and-mouth disease virus A (口蹄疫ウイルス) |
| 分子量 | 理論値: 24.575779 KDa |
| 配列 | 文字列: DKKTEETTLL EDRTLTTRNG HTTSTTQSSV GVTYGYSTGE DHVSGPNTSG LETRVTQAER FFKKHLFNWT TDKPFGHLEK LKLPTDHKG VYGHLVDSFA YMRNGWDVEV SAVGNQFNGG CLLVAMVPEW KKFTPREKYQ LTLFPHQFIS PRTNMTAHIT V PYLGVNRY ...文字列: DKKTEETTLL EDRTLTTRNG HTTSTTQSSV GVTYGYSTGE DHVSGPNTSG LETRVTQAER FFKKHLFNWT TDKPFGHLEK LKLPTDHKG VYGHLVDSFA YMRNGWDVEV SAVGNQFNGG CLLVAMVPEW KKFTPREKYQ LTLFPHQFIS PRTNMTAHIT V PYLGVNRY DQYKKHKPWT LVVMVVSPLT TSSIGATEIK VYANIAPTHV HVAGELPSKE UniProtKB: Genome polyprotein |
-分子 #5: VP3 of capsid protein
| 分子 | 名称: VP3 of capsid protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus A (口蹄疫ウイルス) Foot-and-mouth disease virus A (口蹄疫ウイルス) |
| 分子量 | 理論値: 24.284121 KDa |
| 配列 | 文字列: GIVPVACSDG YGGLVTTDPK TADPVYGKVY NPPRTNYPGR FTNLLDVAEA CPTFLCFDDG KPYVVTREDE QRLLAKFDVS LAAKHMSNT YLSGIAQYYA QYSGTINLHF MFTGSTDSKA RYMVAYVPPG VETPPDTPER AAHCIHAEWD TGLNSKFTFS I PYVSAADY ...文字列: GIVPVACSDG YGGLVTTDPK TADPVYGKVY NPPRTNYPGR FTNLLDVAEA CPTFLCFDDG KPYVVTREDE QRLLAKFDVS LAAKHMSNT YLSGIAQYYA QYSGTINLHF MFTGSTDSKA RYMVAYVPPG VETPPDTPER AAHCIHAEWD TGLNSKFTFS I PYVSAADY AYTASDVAET TNVQGWVCIY QITHGKAQND TLVVSVSAGK DFELRLPIDP RTQ UniProtKB: Genome polyprotein |
-分子 #6: VP4 of capsid protein
| 分子 | 名称: VP4 of capsid protein / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Foot-and-mouth disease virus A (口蹄疫ウイルス) Foot-and-mouth disease virus A (口蹄疫ウイルス) |
| 分子量 | 理論値: 8.778129 KDa |
| 配列 | 文字列: GAGQSSPATG SQNQSGNTGS IINNYYMQQY QNSMDTQLGD NAISGGSNEG STDTTSTHTT NTQNNDWFSK LASSAFSGLF GALLA UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION 回折 回折最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 34373 |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X